マイクロサテライトの多型によるイネいもち病菌株の識別法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
イネいもち病菌のゲノムDNAに存在するマイクロサテライトの多型を解析することにより、イネいもち病菌の菌株の識別が可能である。
- キーワード: イネいもち病菌、マイクロサテライト、多型解析、菌株識別
- 担当:東北農研・地域基盤研究部・病害管理研究室
- 連絡先:電話019-643-3433、電子メールjsakai@affrc.go.jp
- 区分:東北農業・生産環境、共通基盤・病害虫(病害)
- 分類:科学・参考
背景・ねらい
マイクロサテライトは真核生物のゲノムDNA配列中に存在する2∼5塩基の反復配列であり、その反復数の違い(多型)を利用して、 多くの生物で個体識別マーカーとして使われている。公開されたイネいもち病菌のゲノムDNAの塩基配列情報をもとに、マイクロサテライトの多型を検出する ことにより、イネいもち病菌の菌株の識別法を開発する。
成果の内容・特徴
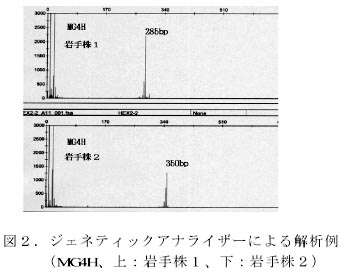
- イネいもち病菌のゲノムDNAの塩基配列情報(Magnaporthe Sequencing Project, http://www.broad.mit.edu)をもとに5種類のプライマーセットを作製した(表1)。これらのプライマーセットを用いてPCR法(図1)によりマイクロサテライトを含む領域を増幅し、得られるDNA断片をジェネティックアナライザー(ABI 310等)で解析することにより、マイクロサテライトの多型を検出する(図2)。
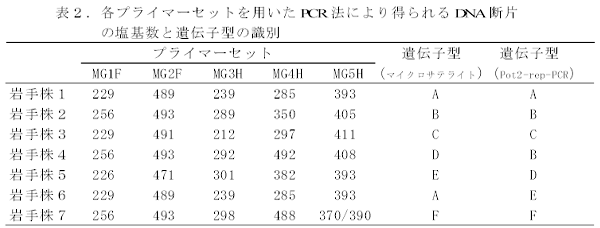
- 隣接する水田から同時期に採取した複数菌株について本識別法を行うと、作製した5種類のプライマーセットにより得られるDNA断片の塩基数から判定した遺伝子型に多型が検出される(表2)。
- 従来行われている菌株識別法であるPot2-rep-PCR法で同一の遺伝子型と判定される2菌株(岩手株2と4)の識別が本法では可能である(表2)。
- 本識別法では、ジェネティックアナライザーによりDNA断片の塩基数を計測することから、同一の基準で遺伝子型の判定ができ、従来の菌株識別法と比べて再現性に優れる。
成果の活用面・留意点
- プライマーセットの分譲は可能である。
- 本識別法は、シミレーションによるイネいもち病菌の個体群動態モデルの検証ならびに薬剤耐性菌の動態の解明に利用できる。
- PCR反応に供試するDNAの調製には市販のキットが利用できる。
- 使用するプライマーセットの数をさらに増やすことにより、より高精度な菌株の識別が可能となる。
- 菌の継代培養等による変異により、遺伝子型が変化する可能性がある。
具体的データ

その他
- 研究課題名: いもち病菌個体群の空間構造のモデル化による遺伝子型頻度の予測
- 課題ID: 05-08-03-02-21-05
- 予算区分: 委託プロ(ブラニチ5系)
- 研究期間: 2003∼2005年度
- 研究担当者: 酒井淳一、兼松誠司、石黒 潔
