ピーマンにおけるPMMoV抵抗性遺伝子(L4)に連鎖したDNAマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
塩基配列が5'-AAGGCGCGAACG-3'のプライマーを用いてトウガラシDNAをPCRすると、PMMoV抵抗性のL4遺伝子と連鎖したDNA断片(WA31-1500)が増幅される。このDNA断片はL4遺伝子を有するPMMoV抵抗性個体選抜のDNAマーカーとして利用できる。
- キーワード:DNAマーカー、PMMoV抵抗性、L4遺伝子、PCR、ピーマン・トウガラシ
- 担当:野菜茶研・果菜研究部・ナス科育種研究室
- 連絡先:059-268-4653
- 区分:野菜茶業・野菜育種
- 分類:科学・参考
背景・ねらい
PMMoV(トウガラシマイルドモットルウイルス)はピーマン栽培において最も重要なウイルス病害であり、効果的な防除法がないため抵抗性品種の育成が望まれている。L3遺伝子を導入した抵抗性品種が既に開発されているが、これら品種を犯すストレインが発生しているため、L3遺伝子より広範囲のストレインに抵抗性を示すL4遺伝子を導入した抵抗性品種の育成が望まれている。しかし、PMMoVは伝染能力が高く、取り扱いに厳重な注意を要し、このことが品種開発に対して大きな妨げとなっている。そこで、PMMoV抵抗性育種における障壁を軽減するため、PMMoV抵抗性のL4遺伝子に連鎖したDNAマーカーを開発する。
成果の内容・特徴
- WA31-1500はトウガラシ近縁種(Capsicum chacoense)由来のL4遺伝子によるPMMoV抵抗性に連鎖したRAPDマーカーである。
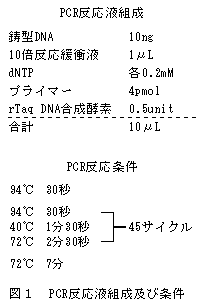
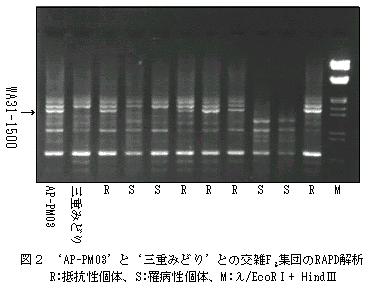
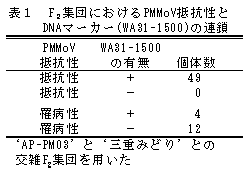
- F2(AP-PM03×三重みどり)から個体別に抽出したDNAを用いて、塩基配列が5'-AAGGCGCGAACG-3'のプライマー(WA31)でPCRをすると(図1)、WA31-1500が増幅された個体は9割以上がPMMoV抵抗性個体である(図2、表1)。
- 21品種・系統のトウガラシ属植物から抽出したDNAを用いてWA31でPCRをすると、WA31-1500が増幅される品種・系統はほとんどがL4遺伝子を有する(表2)。
- WA31-1500はL4遺伝子と密に連鎖しており、L4遺伝子を有するPMMoV抵抗性選抜のためのDNAマーカーとして利用できる。
成果の活用面・留意点
- WA31-1500をDNAマーカーとして用いることによって、PMMoVを栽培施設に持ち込まずに抵抗性個体を絞り込むことができ、ピーマンのPMMoV抵抗性育種を効率的に進めることが出来る。
- WA31-1500は一部の罹病性個体でも増幅されるので、接種検定等による抵抗性の確認が必要である。
具体的データ

その他
- 研究課題名:ピーマンPMMoV抵抗性品種の育成および効率的選抜技術の開発
- 予算区分:連携実用化
- 研究期間:2000~2001年度
- 研究担当者:松永啓、齊藤猛雄、吉田建実
- 発表論文等:松永ら(2001)園学雑70別2:398.
