ピーマンにおけるPMMoV抵抗性遺伝子(L4)に連鎖したSCARマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
トウガラシDNAを20merのプライマーセット(AP-7/8)でPCRをすると、PMMoV抵抗性のL4遺伝子と密に連鎖したDNA断片(WA31-1500S)が増幅される。このDNA断片はL4遺伝子を有するPMMoV抵抗性個体選抜のDNAマーカーとして利用できる。
- キーワード:SCARマーカー、PMMoV抵抗性、L4遺伝子、PCR、ピーマン、トウガラシ
- 担当:野菜茶研・果菜研究部・ナス科育種研究室
- 連絡先:電話059-268-4653、電子メールtatemi@affrc.go.jp
- 区分:野菜茶業・野菜育種
- 分類:科学・参考
背景・ねらい
PMMoV(トウガラシマイルドモットルウイルス)はピーマン栽培において最も重要なウイルス病害であり効果的な防除法がない。そのため、広範囲のストレインに抵抗性を示すL4遺伝子を導入した抵抗性品種の育成が望まれている。しかし、PMMoVは伝染能力が高く、取り扱いに厳重な注意を要し、このことが品種開発に対して大きな妨げとなっている。PMMoV抵抗性育種の効率化のため、これまでに、L4遺伝子に連鎖したDNAマーカー(WA31-1500)を開発したが、抵抗性として選抜した個体群に罹病性個体が含まれていた。そこで、さらに精度が高いDNAマーカーを開発する。
成果の内容・特徴
- WA31-1500Sはトウガラシ近縁種(Capsicum chacoense)由来のL4遺伝子によるPMMoV抵抗性に連鎖したSCAR(sequence characterized amplified region)マーカーである。
- RAPDマーカー(WA31-1500)の塩基配列を基に20merのプライマーセット(AP-7/8)を設計した。L4遺伝子を有する抵抗性系統のAP-PM03及びAP-PM04を片親としたF2集団から個体別に抽出したDNAを用いて、AP-7/8でPCRをすると(図1)、WA31-1500Sが増幅された個体は全てPMMoV抵抗性個体である(表1)。
- 21品種・系統のトウガラシ属植物から抽出したDNAを用いてAP-7/8でPCRをすると、WA31-1500Sが増幅されるすべての品種・系統はL4遺伝子を有する(表2)。
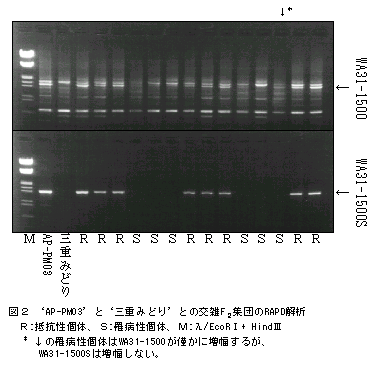
- WA31-1500は一部の罹病性個体で増幅したが、WA31-1500Sは全ての罹病性個体で増幅しない(図2)。
- WA31-1500SはL4遺伝子と密に連鎖しており、L4遺伝子を有するPMMoV抵抗性選抜のためのより有効なSCARマーカーとして利用できる。
成果の活用面・留意点
- WA31-1500Sを選抜マーカーとして用いることによって、PMMoVを栽培施設に持ち込まずに抵抗性個体を絞り込むことができ、ピーマンのPMMoV抵抗性育種を効率的に進めることが出来る。
具体的データ



その他
- 研究課題名:ピーマンPMMoV抵抗性品種の育成および効率的選抜技術の開発
- 予算区分:連携実用化
- 研究期間:2000~2002年
- 研究担当者:松永啓、斎藤 新、齊藤猛雄、吉田建実
