単純反復配列(SSR)マーカーを利用したハクサイ連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
ハクサイにおいて開発された単純反復配列(SSR)マーカーはアブラナ科野菜一般に高い汎用性を持ち、これをランドマークとして基本染色体数と同じ10連鎖群に収束したハクサイの遺伝連鎖地図を構築できる。
- キーワード:ハクサイ、単純反復配列、SSR、遺伝連鎖地図、分子マーカー
- 担当:野菜茶研・機能解析部・育種工学研究室
- 連絡先:電話059-268-4651、電子メールh.fukuoka@affrc.go.jp
- 区分:野菜茶業・野菜育種
- 分類:科学・普及
背景・ねらい
有用形質の遺伝解析やマーカー選抜法開発をはじめとするゲノム情報を利用した育種手法の高度化のためには、汎用性が高く精 度の高い分子マーカーの開発とこれを用いた遺伝連鎖地図の構築が重要な基礎となる。アブラナ科野菜の重要形質の詳細な遺伝解析とその育種利用を進めるため の基本的ゲノム情報を整備するため、共優性であり汎用性の高いSSRマーカーを開発し、これをランドマークとする遺伝連鎖地図を構築する。
成果の内容・特徴
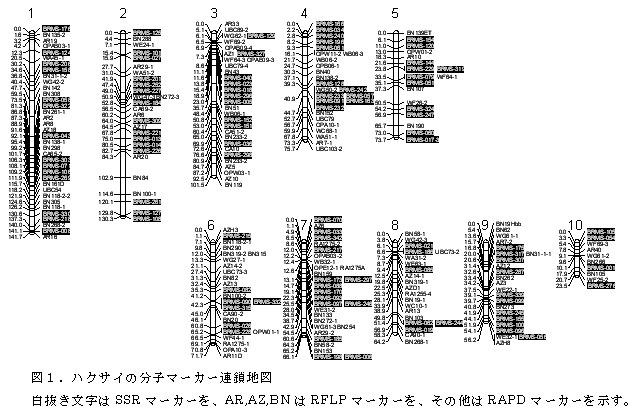
- 'はくさい中間母本農7号'と実験系統'G004'との交雑第2世代94個体を用いた連鎖解析によって構築された遺伝連鎖地図は、ハクサイの基本染色体数と同じ10連鎖群から構成される(図1)。
- この連鎖地図は全長805cMであり、105のSSR、77のRFLP、59のRAPDの合計241マーカーが座乗し、平均マーカー間距離は3.3cMである。
- ハクサイでは上記を含めて361のSSR領域がSTS化されており、その系統間多型はPCRによって検出可能である(表1)。
- 開発されたSSRプライマーは、その多くがキャベツ、ダイコンなどの別種のアブラナ科野菜にも利用でき、モデル植物であるシロイヌナズナに対しても約40%のプライマーが適用可能である(表2)。
成果の活用面・留意点
この知見は、ハクサイをはじめとするアブラナ科野菜の遺伝解析やマーカー育種法開発に広く利用可能である。
具体的データ


その他
- 研究課題名:アブラナ科野菜における連鎖地図の作製と精密化
- 課題ID:11-09-04-01-01-03
- 予算区分:地域バイテク型・重点支援
- 研究期間:1999~2003年度
- 研究担当者:諏訪部圭太、近藤正敏、藤村みゆき、布目司、大山暁男、塚崎光、畠山勝徳、池谷裕幸、釘貫靖久、鈴木徹、平井正志、松元哲、福岡浩之
- 発表論文等:Suwabe et al. (2002) Theor. Appl. Genet. 104:1092-1098.
