単純反復配列マーカー開発法の効率化
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
ゲノムDNAから単純反復配列(SSR、マイクロサテライト)を選択的にクローニングしてライブラリーを構築する方法およびその塩基配列を自動処理してマーカー化のためのPCRプライマーを設計するプログラムによって、単純反復配列を利用した効率的なDNAマーカー作出ができる。
- キーワード:単純反復配列、SSR、マイクロサテライト、濃縮ライブラリー、情報処理
- 担当:野菜茶研・機能解析部・育種工学研究室
- 連絡先:電話059-268-4651、電子メールh.fukuoka@affrc.go.jp
- 区分:野菜茶業・野菜育種、作物・生物工学
- 分類:科学・参考
背景・ねらい
検出操作が簡便で対立遺伝子数が多く共優性に遺伝するという特徴をもつ単純反復配列(SSR)は各種野菜の汎用的なDNAマーカー作出のために利用価値が高い。有用形質の遺伝解析やマーカー選抜法開発をはじめとするゲノム情報を利用した育種手法の高度化のためSSRを各種作物から効率的に単離しDNAマーカー化するための一連の技術を開発し、プロトコール化する。
成果の内容・特徴
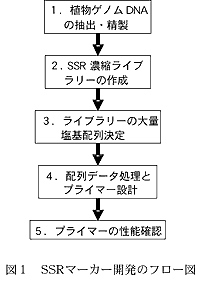
- SSRマーカー開発の流れは第1図に示す。ゲノムDNAのうちSSRを含む断片をビオチン化プローブ(GA)nもしくは(GT)nに結合させて選択し、ベクターにクローニングして濃縮ライブラリーを作成する。その後、無作為に塩基配列決定を行いSSRを含む領域をSTS化する。
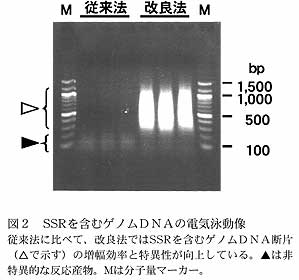
- 断片化したゲノムDNAへ付加するリンカーの配列の設計にあたり、付加反応時に生じる自己2量体を制限酵素XmnIで除去できるように工夫することによって、濃縮ライブラリー作成の際の反応の特異性と効率を著しく向上できる(図2)。
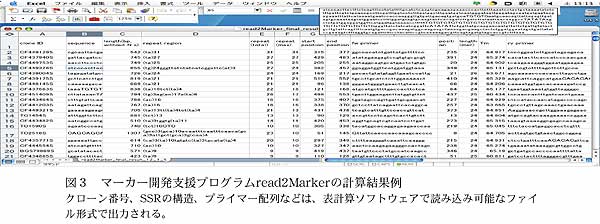
- クローニングされたSSR領域を含むゲノム断片の塩基配列は市販のDNAシーケンサによって決定する。DNAシーケンサからの出力ファイルを自動解析するために開発したプログラムread2Markerにより、SSR領域の検出、クローンの重複や多重遺伝子族と示唆されるものの排除などを自動的に行い、マーカー化のためのPCRプライマーが設計される(図3)。
- ライブラリー構築は特殊な装置を必要とせず、プライマーの自動設計プログラムは一般の市販パーソナルコンピュータ上で動作する。
成果の活用面・留意点
- この成果は、網羅的なゲノム情報に乏しい各種野菜の省力的かつ高精度なDNAマーカー開発に広く利用可能である。
- 本手法はナス、ネギ、ヤマノイモでの実績があり、作物種は特に問わないと考えられる。
- ライブラリー構築法の詳細なマニュアルおよびマーカー設計プログラムとドキュメントファイルはホームページ等で公開する予定である。
具体的データ
その他
- 研究課題名:野菜の高精度DNAマーカーの効率的作出法の開発
- 課題ID:11-09-04-01-14-04
- 予算区分:DNAマーカー
- 研究期間:2002~2004年度
- 研究担当者:布目 司、宮武宏治、大山暁男、河野いづみ(STAFF研究所)、並木信和(STAFF研究所)、福岡浩之
- 発表論文等:1) 布目ら (2004) 育種学研究6(別2):83.
2) 福岡ら (2004) 育種学研究6(別2):84.
