単純反復配列マーカーを利用したナス連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
構築したナスの連鎖地図は、単純反復配列(SSR、マイクロサテライト)マーカーを含む220個のマーカーから構築され、基本染色体数と同じ12連鎖群に収束している。これらの単純反復配列マーカーはナス系統間において高い多型性を有し、ランドマークとして利用できる。
- キーワード:ナス、単純反復配列、SSR、マイクロサテライト、連鎖地図
- 担当:野菜茶研・機能解析部・育種工学研究室
- 連絡先:電話059-268-4651、電子メールnunome@affrc.go.jp
- 区分:野菜茶業・野菜育種、作物・生物工学
- 分類:科学・参考
背景・ねらい
有用形質の遺伝解析やマーカー選抜法開発をはじめとするゲノム情報を利用した育種法の高度化のためには、汎用性が高く精度の高い分子マーカーの開発とこれを用いた遺伝連鎖地図の構築が重要な基礎となる。ナスの重要形質の詳細な遺伝解析とその育種利用を計るための基本的ゲノム情報を整備するため、共優性を示し汎用性の高いSSRマーカーを開発し、これをランドマークとする詳細な遺伝連鎖地図を構築する。
成果の内容・特徴
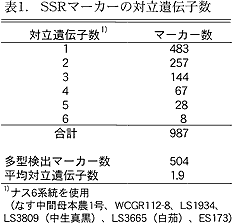
- 「なす中間母本農1号」よりSSR濃縮ライブラリを作成し、その塩基配列情報から987のナスSSRをSTS化した。そのうち504個は、ナス6系統間で多型を少なくとも1つ検出できる(表1)。
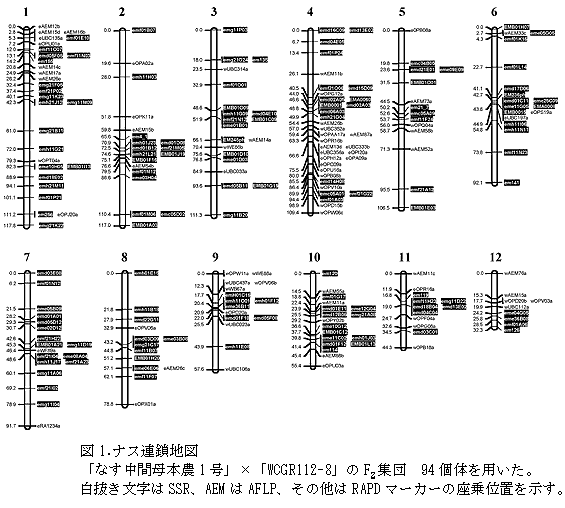
- 「なす中間母本農1号」と育種素材系統「WCGR112-8」との交雑第2世代94個体を用いた連鎖解析により作成した遺伝連鎖地図は、ナスの基本染色体数と同じ12連鎖群から構成される(図1)。
- この連鎖地図は全長1016cMであり、147個のSSR、23個のAFLP、50個のRAPD、合計220個のマーカーが座乗し、平均マーカー間距離は4.9cMである(図1)。
- 開発されたSSRマーカーは、異なる交配組み合わせを用いた遺伝解析にも利用可能である(表2)。
成果の活用面・留意点
- 作成された連鎖地図はナスの詳細な遺伝解析やマーカー選抜法開発に利用できる。
- 開発したSSRマーカーは、ナス系統間の多型をPCRにより検出でき、ナス品種系統識別に利用可能である。
具体的データ

その他
- 研究課題名:野菜の高精度DNAマーカーの効率的作出法の開発
- 課題ID:11-09-04-01-14-04
- 予算区分:DNAマーカー
- 研究期間:2002~2004年度
- 研究担当者:布目 司、諏訪部圭太、宮武宏治、大山暁男、福岡浩之
- 発表論文等:1) Nunome et al. (2004) XIIth Eucarpia meeting on genetics and breeding of Capsicum and
eggplant. p.251.
2) 布目ら (2004) 育種学研究6(別2):83
