ハクサイ根こぶ病抵抗性遺伝子座近傍の詳細連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
ハクサイとシロイヌナズナのシンテニー情報を利用して、ハクサイ根こぶ病抵抗性遺伝子座の詳細連鎖地図を効率的に作成することができる。開発したマーカーBSA7と抵抗性遺伝子座Crr1 は極めて強い連鎖関係にある。
- キーワード:ハクサイ、シロイヌナズナ、根こぶ病抵抗性、シンテニー
- 担当:野菜茶研・機能解析部・野菜ゲノム研究チーム
- 連絡先:電話050-3533-3863、電子メールssmats@affrc.go.jp
- 区分:野菜茶業・野菜育種、作物・生物工学
- 分類:科学・参考
背景・ねらい
アブラナ科野菜にとって根こぶ病は難防除土壌病害の一つであり、抵抗性個体の効率的な選抜法の開発や耕種的防除につながる抵抗性機構の解明が望まれている。そのため抵抗性遺伝子座近傍の詳細な連鎖地図を作成し抵抗性遺伝子を単離することにより、発現機構の解析に結び付けることが重要である。そこでシロイヌナズナとのシンテニー解析により、2個のハクサイ根こぶ病抵抗性遺伝子座(Crr1、Crr2)周辺のマーカーを効率的に開発し、抵抗性に強く寄与しているCrr1 近傍について詳細連鎖地図を作成する。
成果の内容・特徴
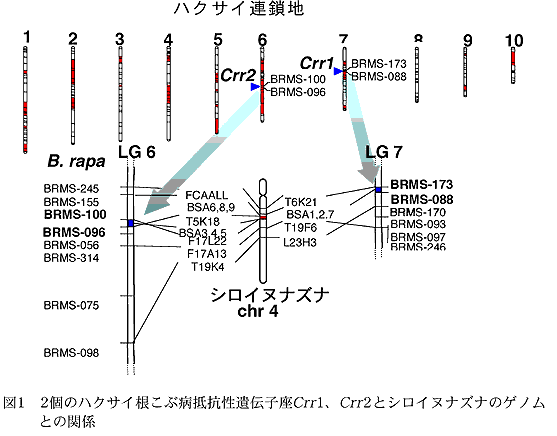
- Crr1 とCrr2 に連鎖するSSRマーカー配列は、いずれも同じシロイヌナズナの第4染色体の病害抵抗性遺伝子がクラスターを形成している領域と高い相同性が認められる(図1)。Crr1 とCrr2 とは同じ起源の抵抗性遺伝子であると推定される。
- 相同性が認められるシロイヌナズナの周辺配列情報を利用して、新たに得られた連鎖マーカーは、Crr1 では4個(BSA1、BSA2、AT27、BSA7)、Crr2 では3個(BSA3、BSA4、BSA5)である。これらは罹病性親と抵抗性親間で見つかった一塩基多型または一塩基多型の周辺領域で検出した挿入・欠失配列に基づくマーカーである。
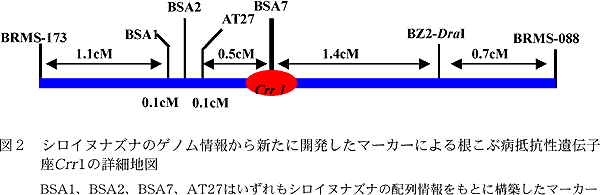
- Crr1 に連鎖する5個のマーカーは、2個のSSRマーカー(BRMS-173、BRMS-088)約4cM間に0.1~1.4cM間隔で(図2)、またCrr2 に連鎖する5個のマーカーは、4.9cM間に0.2~1.9cM間隔で座乗する。
- Crr1 とBSA7は、AT27とBZ2-Dra I間に位置しており、解析したF2 1920個体からなる集団では組換え個体が得られず、極めて強い連鎖関係にある(図2)。
成果の活用面・留意点
- 本連鎖地図は、抵抗性遺伝子の単離に有用な情報となる。
具体的データ
その他
- 研究課題名:ハクサイ根こぶ病抵抗性遺伝子の単離
- 課題ID:11-09-04-01-23-04
- 予算区分:重点研究課題
- 研究期間:2004年度
- 研究担当者:松元 哲、諏訪部圭太、藤村みゆき、近藤正敏、國久美由紀、布目 司、福岡浩之、
佐藤隆徳、小島昭夫 - 発表論文等:1) Suwabe et al (2004) Brassica 2004 Abstract Book pp.143
2) 松元ら (2004) 育種学研究 6(1), p79
