染色体に対応付けられた単純反復配列(SSR)を基にしたネギ連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
約250個のネギ単純反復配列(SSR)マーカーおよびタマネギ発現遺伝子配列タグ(EST)マーカーで構築された連鎖地図は、17連鎖群、全長2116cMである。これらの連鎖群は、ネギおよびタマネギの染色体と対応付けられている。
- キーワード:ネギ、連鎖地図、DNAマーカー、マイクロサテライト、染色体、タマネギ
- 担当:野菜茶研・野菜育種研究チーム
- 代表連絡先:電話050-3533-3863
- 区分:野菜茶業・野菜育種
- 分類:研究・普及
背景・ねらい
有用形質の遺伝様式を明らかにするためには、連鎖地図を用いたQTL解析が有効である。また、汎用性の高いDNAマーカーを利用した連鎖地図を構築することにより、近縁種とのシンテニー(異なる種の染色体間で遺伝子の配列が類似していること)を利用した有用形質のマッピングも可能となる。しかし、ネギ属野菜ではゲノム情報の蓄積が乏しく、DNAマーカーの開発が遅れており、遺伝子型の識別能が高い共優性マーカーを主体とした連鎖地図は構築されていない。そこで、ネギおよびタマネギの遺伝情報から、共優性を示し、汎用性の高いDNAマーカーを基にしたネギ連鎖地図を構築する。
成果の内容・特徴
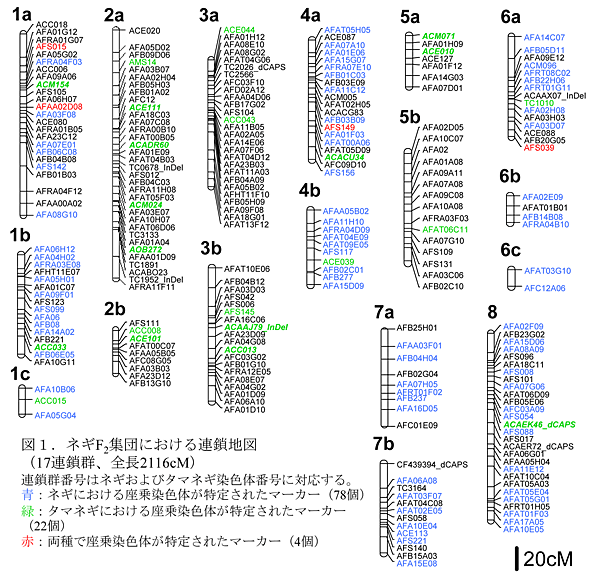
- ネギ千住群品種「西光」由来自殖系統D1s-15s-10sと九条群品種「九条太」由来自殖系統J1s-14s-23sとのF2集団225個体を基に構築した連鎖地図は、17連鎖群からなり、全長2116cMである(図1)。
- この連鎖地図には、ネギSSR 213個、タマネギEST 41個(うちEST-SSR 16個)、タマネギゲノム由来SSR 1個の合計255個のマーカーが座乗する(図1)。
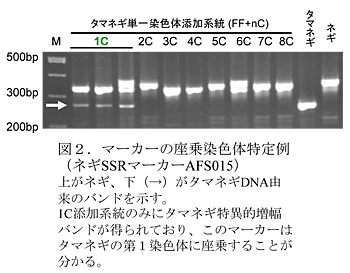
- タマネギ染色体を1本添加したネギ系統(タマネギ単一染色体添加系統、ゲノム構成FF+nC、2n=17)のシリーズ(FF+1C~+8C)およびネギ染色体を1本欠失したタマネギとネギの二基三倍体(ネギ単一染色体欠失系統、ゲノム構成CCF-nF、2n=23)5系統(CCF-1F、-4F、-6F、-7F、-8F)を用いた解析により、17連鎖群全てがネギまたはタマネギの8本の染色体と対応付けられている(図1、図2)。
成果の活用面・留意点
- 連鎖地図の作成に用いたSSRマーカーは多型性が高く、また共優性を示すことから、異なる交配組み合わせを用いた遺伝解析や系統分類にも利用可能である。
- タマネギ(Allium cepa L.)における座乗染色体特定には、シャロット(A. cepa L. Aggregatum Group)およびシャロットとネギ(A. fistulosum L.)との種間雑種に由来する系統を用いた。
- この連鎖地図には、タマネギ連鎖地図(Martinら2005, Mol.Gen.Genet.274: 197-204)と共通するマーカーが13個座乗する(図1において太字の斜体緑字で示す)。
- ネギ連鎖地図および座乗するSSRマーカーに関する情報は、野菜DNAデータベース(VegMarks,http://vegmarks.nivot.affrc.go.jp/VegMarks/jsp/index.jsp)で公開されている。
具体的データ
その他
- 研究課題名:病虫害抵抗性、省力・機械化適性、良食味等を有する野菜品種の育成
- 課題ID:211-j
- 予算区分:委託プロ(グリーンテクノ)、科研費、交付金プロ(園芸マーカ-)、基盤研究費
- 研究期間:2004~2008 年度
- 研究担当者:塚崎光、山下謙一郎、小原隆由、布目司、福岡浩之、米丸淳一(生物研)、
金森裕之(STAFF 研)、河野いづみ(STAFF 研)、執行正義(山口大農)、小島昭夫、若生忠幸 - 発表論文等:1)Tsukazaki H. et al. (2007) Euphytica 157(1-2): 83-94
2)Tsukazaki H. et al. (2008) Theor. Appl. Genet. 117(8): 1213-1223
