Tm-shiftタイピング法に基づく一塩基多型マーカー設計の標準工程
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
一塩基多型(SNPs)の安価かつ省力的なマーカー化手法の1つであるTm-shift タイピング法において、反応条件の検討手順の最適化と専用のプライマー設計プログラムの開発によって確立した標準工程に従うと、80%を越えるマーカー化成功率が得られる。
- キーワード:一塩基多型、SNPs、DNAマーカー、タイピング、Tm-shift、標準工程
- 担当:野菜茶研・野菜ゲノム研究チーム
- 代表連絡先:電話050-3533-3863
- 区分:野菜茶業・野菜育種
- 分類:研究・参考
背景・ねらい
ゲノム全体に高い多型頻度が期待できる一塩基多型(SNPs)の利用は、DNA多型頻度の低い野菜の品種や育種母本を用いたDNAマーカー開発のために重要である。SNPsのマーカー化技術は数多く報告されているが、なかでも安価かつ省力的な手法として期待されるTm-shift タイピング法の反応条件を検討し、それぞれの条件におけるマーカー化成功率に基づいて検討コストの最小化を図るとともに、最適なプライマーの自動設計プログラムを開発し、SNPsマーカー開発の標準工程を確立する。
成果の内容・特徴
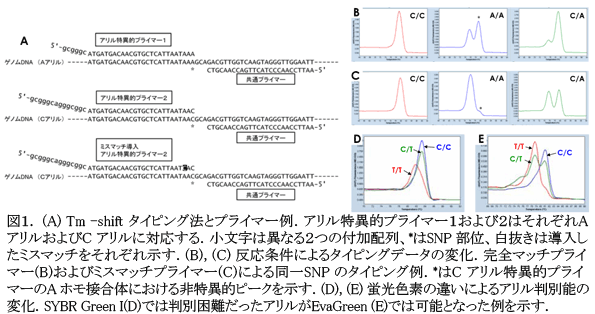
- Tm-shiftタイピング法はSNPsをタイピングする一手法である。それぞれのアリル特異的プライマーに異なる長さの配列を付加して増幅産物の融解温度(Tm)の差異を拡大し、リアルタイムPCR装置を用いて遺伝子型を判定する (図1A)。原報(Wang et al., 2005)では特殊な改変型酵素が不可欠であり、汎用性に問題が残る。
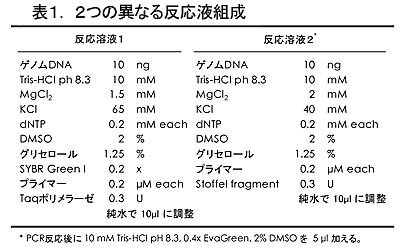
- 一般に入手可能な酵素を用いても、異なる蛍光色素と増幅酵素についてそれぞれ最適化した反応溶液(表1)とアリル特異的プライマーへのミスマッチ導入の有無(図1A)とを組み合わせて安定なタイピングが可能であるが、個々のマーカーごとに反応条件の検討が必要である(図1B-E)。
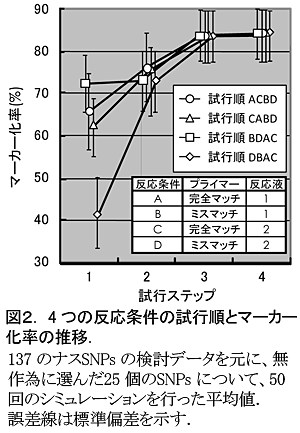
- 137のナスSNPsを用いた実験データに基づくシミュレーションから、ミスマッチ導入プライマーと反応溶液1を組み合わせた場合に最も高い成功率(平均73%)を期待できるが、それ以外の条件が適するSNPs座も存在する(図2)。
- 反応溶液1とミスマッチプライマーを用いて最初の試行を行い、マーカー化できなかった座位だけを対象に反応溶液1および2と完全マッチプライマーによる再検討を行えば、最も効率的に80%以上のSNPsをマーカー化できる。他の検討手順では、必要な条件設定実験数およびプライマー合成コストは増加する(図2)。
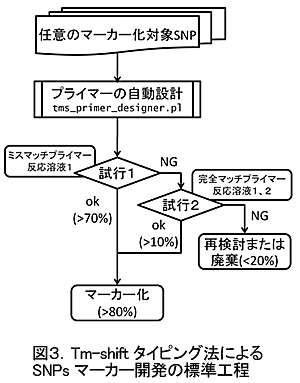
- 最適なプライマーセットを自動的に設計するために独自開発したプログラム「tms_primer_designer」を併用し、図3に示す標準工程に準拠して開発を行うことにより、試行錯誤をせず効率的なSNPsマーカーの作出が可能である。
成果の活用面・留意点
- 本工程に従い、トマトおよびその近縁野生種Solanum pennellii、ピーマンなどにおいても同様の効率が得られている。電気泳動分析や蛍光プライマーを用いない安価で省力的なSNPsマーカーの作出法として、生物種を問わず広く利用可能である。
- Tm-shiftタイピング法の原報およびミスマッチの導入ルールは、それぞれWang et al.(BioTechniques 39:885-893.)、Hayashi et al. (TAG. 108:1212-1220.)を参照のこと。
- 詳細なマニュアルおよびプライマー設計プログラム「tms_primer_designer」は上記の代表連絡先から入手できる。ホームページからの公開も近く行う予定である。
具体的データ
その他
- 研究課題名:野菜におけるDNAマーカー整備及び遺伝子機能解明と利用技術の開発
- 課題ID:221-i
- 予算区分:委託プロ(新農業展開プロ)、交付金プロ(園芸マーカー)
- 研究期間:2007~2008年度
- 研究担当者:福岡浩之、宮武宏治、根来里美、布目司、大山暁男、山口博隆
- 発表論文等:Fukuoka H. et al. (2008) Breed. Sci. 58(4):461-464.
