ナスにおける発現遺伝子配列の大量解読とカタログ化
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
ナスの様々な組織から作製した21種類のcDNAライブラリーから解読した60,000個を超えるクローンの塩基配列情報は16,245種の独立な遺伝子配列に整理され、ナス属(Solanum)植物の新たな網羅的発現遺伝子情報を提供する。
- キーワード:ナス、EST、発現遺伝子配列、データベース
- 担当:野菜茶研・野菜ゲノム研究チーム
- 代表連絡先:電話050-3533-3863
- 区分:野菜茶業・野菜育種
- 分類:研究・普及
背景・ねらい
ナス(Solanum melongena L.)は我が国およびアジア・中近東諸国では最も重要な野菜の1つであるが、分子遺伝学的基盤情報の整備は同じナス科ナス属のトマト(S. lycopersicum L.)やジャガイモ(S. tuberosum L.)と比較して大きく遅れている。そこで、これらのモデル作物との比較ゲノム学的解析による効率的DNAマーカー開発や網羅的遺伝子発現解析による重要形質の機能解析等、ゲノム情報を利用した分子遺伝生理学的アプローチによるナスの育種技術開発を可能とする基盤情報の整備を目的として、ナス発現遺伝子配列(EST)の大量解読とカタログ化を行う。
成果の内容・特徴
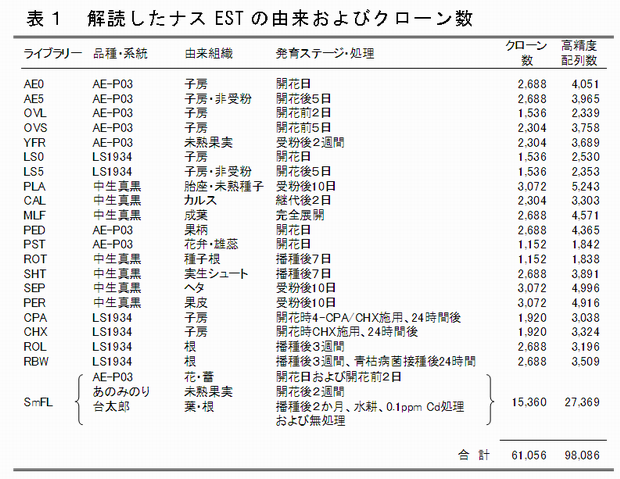
- ナスの様々な品種・系統、組織、発生ステージ・処理に由来する61,056個のcDNAクローンの両末端からサンガー法によって解読された高精度配列数は98,086リードである(表1)。その塩基配列情報の全塩基数は50,438,137bpとこれまでに蓄積されている情報(3,181リード、1,169,411bp)の40倍以上の情報量である。
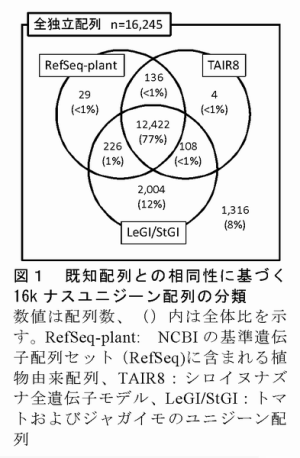
- 得られた塩基配列情報を整理統合しカタログ化すると、16,245種類の独立配列のデータセット(16kナスユニジーンセット)が得られる。既知配列との相同性解析結果は、全体の約77% (12,422配列)が植物全体に広く共通する遺伝子である一方、約12% (2,004配列)はナス科植物のみ、約8%(1,316配列)はナスのみに特徴的な遺伝子であることを示している(図1)。
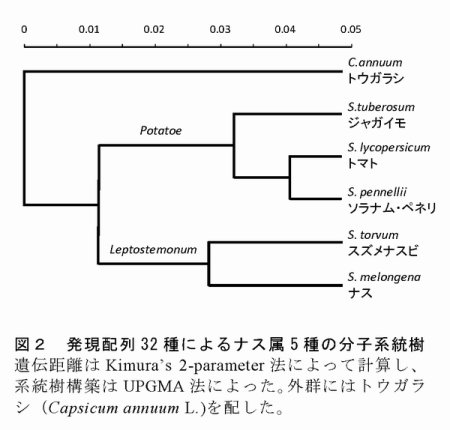
- ナス属のモデル植物であるトマトおよびジャガイモはいずれもPotatoe亜属の新世界起源種であるが、ナスはLeptostemonum亜属の旧世界起源種であるとされており、発現遺伝子配列に基づく分子系統樹において両者は明瞭に区別されたクラスタを形成する(図2)。16kナスユニジーンセットは、ナス属の比較ゲノム学においてナスをトマト、ジャガイモに次ぐ新たな第3極に位置づけ得るデータセットとして利用できる。
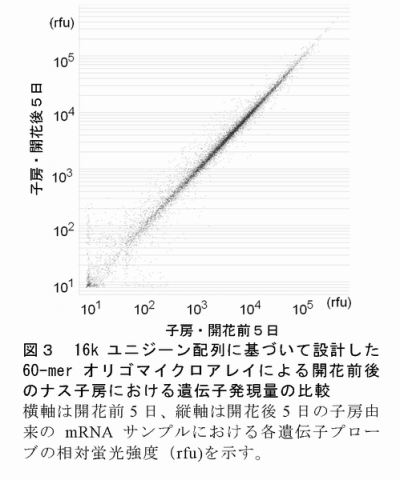
- 得られた配列情報に基づいて開発されたマイクロアレイは遺伝子発現解析に必要十分な再現性と検出感度を有し(図3)、ナスの網羅的遺伝子解析ツールとして利用できる。
成果の活用面・留意点
- 全ての発現配列情報は国際塩基配列データベース(DDBJ/EMBL/GenBank)より登録番号FS000001-FS098086として個別に公開されている。以下のURLからの一括取得も可能である(ftp://ftp.ddbj.nig.ac.jp/ddbj_database/mass/Solanum_melongena_EST)。
- 16kユニジーンセットの配列情報や既知配列との相同性に基づいて付与された推定機能情報は野菜茶研のデータベース「VegMarks」からダウンロードすることができる。(http://vegmarks.nivot.affrc.go.jp/VegMarks/jsp/page.do~transition=link)
具体的データ
その他
- 研究課題名:野菜におけるDNAマーカー整備及び遺伝子機能解明と利用技術の開発
- 中課題整理番号:221i
- 予算区分:新技術・新分野創出、交付金プロ(結実性向上)
- 研究期間:2006~2009年度
- 研究担当者:福岡浩之、山口博隆、布目司、根来里美、宮武宏治、大山暁男
- 発表論文等:Fukuoka H. et al. (2010) Gene 450: 76-84
