遺伝子発現を利用したニラの鮮度評価法
要約
ニラの収穫後に起きる遺伝子発現の変化を明らかにして、貯蔵に伴って発現が特徴的に変動する遺伝子を特定し、RT-PCRで半定量的に検出することにより、ニラの収穫後の鮮度評価を行うことができる。
- キーワード:鮮度評価、遺伝子発現、RT-PCR、マルチプレックス、ニラ、黄化
- 担当:野菜茶研・野菜・茶の食味食感・安全性研究チーム
- 代表連絡先:電話050-3533-3863
- 区分:野菜茶業・野菜品質・機能性
- 分類:研究・参考
背景・ねらい
消費者が野菜を購入する際に鮮度は重要な評価項目である。しかし、現状では野菜の鮮度の定義や評価法は定まっていない。多くの場合、鮮度は外観や物性、品質成分の変化等で評価されるが、栽培時期によって収穫時の成分含量や物性値は一定ではないため、野菜の生理状態を反映した鮮度評価は困難である。
野菜の流通や貯蔵に伴い、切断傷害や流通中の水分ストレス、暗黒ストレスなどによって様々な生理変化が起こる。この過程では、まず遺伝子が発現し、それが酵素活性の変化となって成分含量や物性が変化していく。そこで、収穫後に特異的な発現変化を示す遺伝子を特定するとともに、これらの遺伝子を鮮度のマーカーとして利用して鮮度低下を評価できる方法を開発する。さらに、PCR装置を有する研究施設であればどこでも鮮度評価が実施できるように、マルチプレックスRT-PCR化する。
成果の内容・特徴
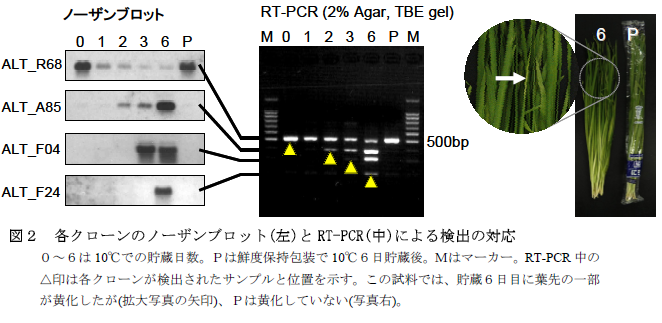
- ニラを10°Cで貯蔵して6日後に葉先の一部に黄化が認められた試料と、貯蔵前の試料からRNAを抽出して、サブトラクション法で発現量の異なる遺伝子を濃縮し、塩基配列が明らかになったのは216クローンである。
- 他の植物種の既知遺伝子と類似性のある27個の遺伝子について、ノーザンブロット法により貯蔵に伴う発現変化を調べ、それぞれの遺伝子の増減のタイミングによりグループ分けする。各グループから1つずつ遺伝子を選び、RT-PCRによってそれらを検出するプライマーを設計する。
- さらに、各遺伝子の発現を、1本のPCR反応で異なった位置に検出できるようにプライマーの塩基配列(表1)や濃度等を調整すると、マルチプレックスRT-PCRによって、半定量的に鮮度低下の進行を簡易に可視化することができる(図1)。
- この方法を利用して、外観に黄化が見られる前に、鮮度低下の兆候をALT_A85、ALT_F04クローンの発現として検出できる(図2)。鮮度保持包装により黄化が抑制されているニラでは、これらのクローンの発現が抑制される。
- RNAの抽出から結果が得られるまでおよそ半日で解析できる。
成果の活用面・留意点
- 本法のプライマーセットはニラの鮮度評価専用であるが、同様の研究手法は他の品目に応用可能である。
具体的データ


その他
- 研究課題名:野菜・茶の食味食感評価法の高度化と高品質流通技術の開発
- 中課題整理番号:311g
- 予算区分:実用技術
- 研究期間:2007~2009年度
- 研究担当者:永田雅靖
- 発表論文等:永田「青果物の鮮度評価方法および鮮度評価用プライマーセット」特願2010-117512
