ピーマンのSSR(単純反復配列)マーカーと高密度連鎖地図
要約
新たに開発したゲノムおよびcDNA配列由来のSSRマーカーを用いて作成したピーマンの高密度連鎖地図は、12連鎖群に収束し、種間交雑F2集団より作成された既知の基本連鎖地図およびトマトゲノム配列と対応付けられている。
- キーワード:ピーマン、連鎖地図、SSR、トマトゲノム
- 担当:日本型施設園芸・野菜ゲノム利用技術
- 代表連絡先:電話 050-3533-3861
- 研究所名:野菜茶業研究所・野菜育種・ゲノム研究領域
- 分類:研究成果情報
背景・ねらい
DNAマーカー利用選抜においては、迅速かつ簡便に利用可能なPCRベースマーカーが強く望まれており、特に品種育成現場では、栽培種内、それも系譜が似る系統同士の交雑も日常的であることから、アリル変異の検出力の大きいSSRマーカーの活用場面が非常に多い。加えて、SSRマーカーは、多系交配集団におけるマルチアリル構成を反映させることも可能であり、遺伝学的にも応用範囲の広いマーカーといえる。しかしながらピーマンにおいては、利用可能なSSRマーカーの情報が非常に限られている。そこで、ピーマンのSSRマーカーを大量に作出し、連鎖地図を介したゲノム上の位置情報も明らかにすることにより、ピーマンにおけるDNAマーカーを利用した選抜や遺伝解析のための基盤情報整備を図る。
成果の内容・特徴
- ピーマンのSSR濃縮ゲノムライブラリー(GAあるいはCA繰り返し配列を対象)から開発したゲノムSSRマーカーの総数は1,736個である。
- 公開cDNA配列データベース(CAGI.release_2)に登録された13,003のピーマンcDNA配列から開発したSSRマーカーの総数は1,344個である。
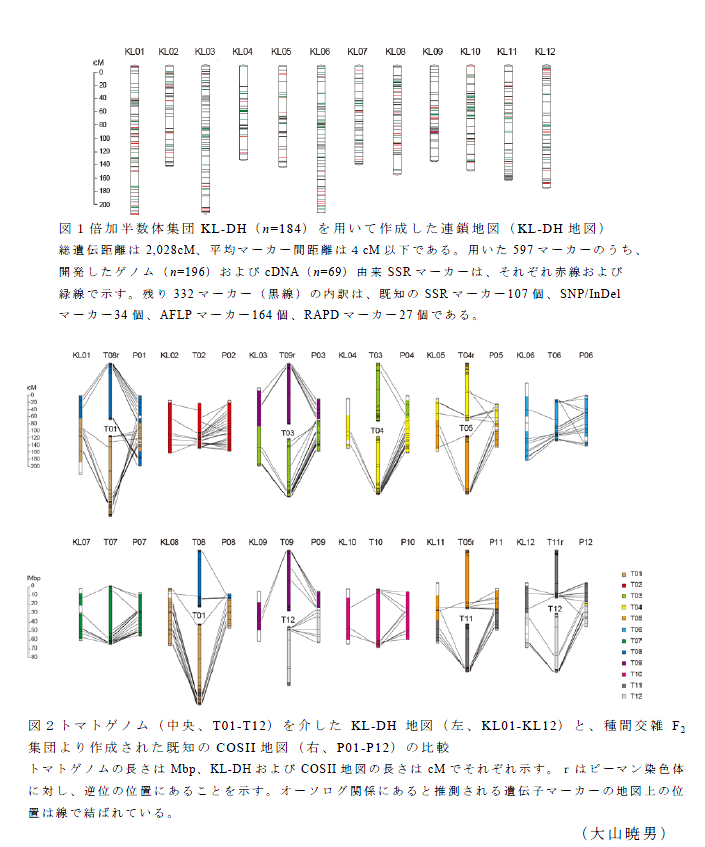
- 開発した3,080個のSSRマーカーのうち、265個を含む計597のDNAマーカーを用い、ピーマン系統K9-11およびMZC-180(宮崎県育成)を両親とするF1小胞子由来の倍加半数体集団KL-DH(n=184)について作成した連鎖地図(KL-DH地図)の連鎖群数は、ピーマンの染色体数12に収束している(図1)。
- KL-DH地図とトマトゲノム配列との対応関係は、種間交雑F2集団より作成された基本連鎖地図(COSII地図、http://solgenomics.net/)の対応関係と類似し、連鎖群(染色体)2、6、7、10番はトマトゲノムと1:1の関係、連鎖群3、5、11、12番は、1:2の関係にある(図2)。
成果の活用面・留意点
- 開発したSSRマーカー情報は、野菜茶業研究所DNAマーカーデータベース(VegMarks)において公開しており、ピーマンの遺伝・QTL解析、マーカー利用選抜、品種・系統識別などに利用できる。
- ピーマン連鎖地図とトマトゲノムとの対応関係は、新たなピーマンのマーカー検索に活用できる。
- マーカーによっては、2塩基程度のDNA断片長の差異を検出できる高精度な解析法を用いる必要がある。
具体的データ
その他
- 中課題名:野菜におけるゲノム情報基盤の構築と利用技術の開発
- 中課題整理番号:141g0
- 予算区分:高度化事業、実用技術
- 研究期間:2006~2012年度
- 研究担当者:大山暁男、福岡浩之、杉田亘(宮崎総農試)、世見由香里(宮崎総農試)、根来里美、宮武宏治、山口博隆、布目司
- 発表論文等:Sugita T. et al. (2013) Mol. Breed. 31:909-920
