RLGS法によるカンキツ遺伝子構造の品種間差の検出
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
代表的なカンキツ10品種についてRLGS解析を行い、枝変りや珠心胚実生の過程で変異したと思われる品種間差を示すスポットを見出した。また、体細胞変異の発生メカニズムを解明する糸口になると考えられる、品種成立の過程で消失と出現を示す特異なスポット(phantom spot)を見出した。
- 担当:四国農業試験場・作物開発部・品質評価研究室
- 連絡先:0877-62-0800
- 部会名:生物工学・果樹
- 専門:生物工学
- 対象:果樹類
- 分類:研究
背景・ねらい
カンキツ栽培では糖度や食味、熟期などにおいて優れた形質を有する品種が求められ、収穫時期や果実品質、栽培適性などの異なる多様な品種の育成・選抜が行われているが、どのような遺伝的、生理的なメカニズムがこれらの形質と関連しているかについての直接的な知見は少ない。
カンキツでは、在来品種の枝変わりに由来する優良系統が多い。これらは少数の遺伝子の変異に由来すると考えられ、これらの微細な遺伝的変異を高感度で特定してその構造を解析し、遺伝的変異と栽培特性との相関を明らかにすることが出来れば、生理特性研究や育種、さらには品種判別技術といった多方面の研究に大きく貢献すると考えられる。本研究では、近年開発されたRLGS法を用いてカンキツの遺伝子構造の微細な変異を特定するための方法を確立する。
成果の内容・特徴
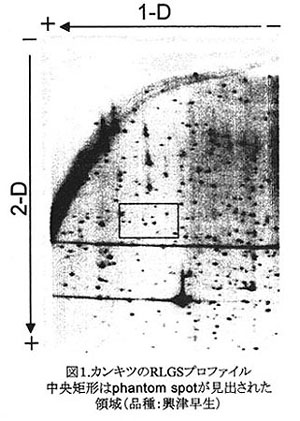
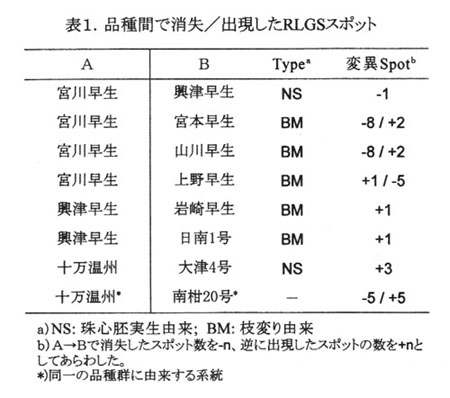
- 四国農試内に栽植されているウンシュウミカン10品種を材料としてRLGS解析による遺伝子構造の解析を行ったところ、特定の塩基配列(NotI配列)を目印とした場合、カンキツでは400個近くのスポットが得られた(図1)。枝変わりと珠心胚実生に由来する品種間でRLGSスポットのパターンを比較したところ各品種間で出現もしくは消失するスポットが見出された(表1)。RLGS法は枝変わりばかりでなく、珠心胚実生で生じた遺伝的に極めて近縁な品種の遺伝子分析法として有効である。
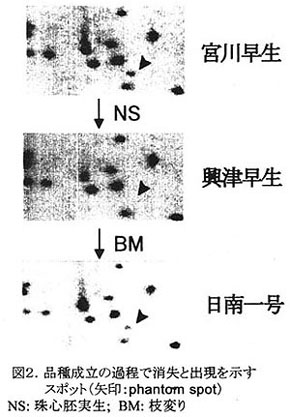
- 変異が繰り返し起こったと考えられる品種系統において、一度消失したものの、再度出現するスポット(phantom spot)を1個見出した。(図2)。
成果の活用面・留意点
- カンキツの枝変わりなどを用いることで、農業上重要な形質に関連していると思われる遺伝子の極めて微細な変化をRLGS法で迅速に検出する可能性が示された。今後、RLGS分析の条件を更に改良し、また変異の認められたスポットをクローニングしてその構造を解析することで、目的形質の発現に直接関係する遺伝子を短期間で特定することが出来ると考えられる。
本年度は1品種1~2樹から抽出したDNAを用いた結果であるので、同一品種内での複数樹を対象として各品種のRLGSプロファイルの一般性を確認する必要がある。
また、Phantom spotの性情を今後詳細に解析する事で、枝変りなどの体細胞変異の発生機構の解明への貢献が期待される。
具体的データ
その他
- 研究課題名:RLGS法によるカンキツ遺伝子構造の品種間差の解析
- 予算区分:経常(場内プロジェクト)
- 研究期間:平成9年度
- 研究担当者:清水徳朗、河瀬真琴
- 発表論文等:RLGS analysis of Citrus unshiu’ Shimizu T and M. Kawase、平成10年度園芸学会春季大会(1998)、他
