小麦低分子グルテニン遺伝子型の簡易判別技術
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
農林61号の低分子グルテニン遺伝子群は12グループ6タイプに分類出来、それらの遺伝子座を同定出来る。また、PCR法により個々の遺伝子を簡易判別することが可能である。
- キーワード:小麦、生地物性、低分子グルテニン、遺伝子座、PCR法、簡易判別
- 担当:農研機構・近中四農研・作物開発部・育種工学研究室
- 連絡先:084-923-4100
- 区分:近畿中国四国農業・生物工学
- 分類:科学・参考
背景・ねらい
小麦粉のタンパク質は主にグルテニンとグリアジンから成っており、小麦粉に水を加えてこねることによりグルテンを形成する。この内、グルテニンの遺伝子型は小麦の生地物性に大きく影響し、その育種的改良はパンやめん用小麦の品質向上に重要である。グルテニンの中でも、低分子グルテニンサブユニット(LMW-GS)については、高分子グルテニンサブユニットと同様に生地物性に対する効果が明らかになっているが、サブユニット数が多く、その遺伝子型のタンパク質レベルでの解析が困難である。そこで、日本品種の製パン性および製めん性の向上のため、LMW-GS遺伝子の構成を明らかにし、その遺伝子型の効率的な選抜を可能にする簡易判別技術の開発を行う。
成果の内容・特徴
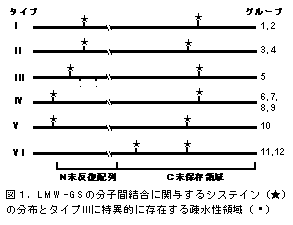
- 農林61号のLMW-GS遺伝子群をクローニングし、その予想される保存領域のアミノ酸配列から、12グループ(1~12)に分けることが出来、さらに、グルテニン分子間結合による網目構造の形成に関わるシステインの分布から、12グループは6つのタイプ(I~VI)に分けることが出来る。
- グループ5(タイプIII)は、他のサブユニットでは親水性のアミノ酸が多い反復配列領域に5つの疎水性アミノ酸からなる反復配列をコードしており、生地物性に対して他のサブユニットとは異なった効果を持つことが予想出来る(図1)。
- グループ11と12(タイプVI)は他のサブユニットと異なり、N末端にシステインを持たないことから、グルテニン分子間結合による網目構造の形成に影響を与えることが予想出来る(図1)。
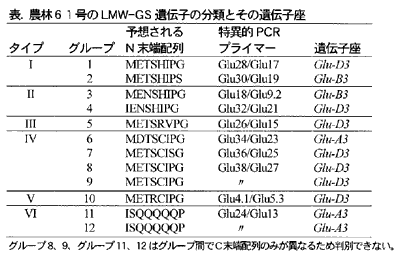
- 12グループのLMW-GS遺伝子を特異的に増幅するPCRプライマーを設計し、他のグループと類似性が高い2グループを除き、各グループ特異的なPCR産物を得ることが出来る(表)。
- 各グループの遺伝子座は、3つのグループがGlu-A3、2つのグループがGlu-B3、7つのグループがGlu-D3に座乗する(表)。
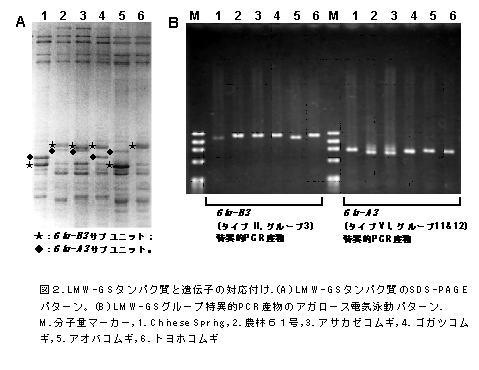
- LMW-GS遺伝子型の品種間差異については、グループ3(タイプII)、グループ11と12(タイプVI)で多型を検出し、それらは対応するタンパク質の多型に対応することから、この方法はLMW-GSの多型の検出に有効である(図2)。
成果の活用面・留意点
- アオバコムギの持つGlu-B3座のLMW-GSは、生地物性の向上に効果があることが明らかになっており、これを検出するグループ3の特異的プライマーは、このサブユニットの簡易選抜に利用可能である。
- PCR法による遺伝子型の検定では、タンパク質として発現していない偽遺伝子を検出してしまう可能性があるので、場合によってはタンパク質レベルでの分析が必要である。
- 今回開発したPCRプライマーで検出できない変異がある可能性があるので、今後、新たなPCRプライマーを開発する必要がある。
- システインを1つ持つ変異型グリアジンがグルテニン分画に存在し、LMW-GSとよく似た分子量持つため、タンパク質電気泳動法で見られるLMW-GSの多型の中にはグリアジン由来のタンパク質も存在する。これら変異型グリアジンを検定するには、新たなPCRプライマーを開発する必要がある。
- 品種系統間の生地物性の差異と対応付けられるLMW-GSの同定を今後行っていく必要がある。
具体的データ
その他
- 研究課題名:小麦生地物性改良のための低分子グルテニン遺伝子型の簡易判別技術の開発
- 予算区分:交付金、21世紀プロジェクト1系
- 研究期間:1998~2001年度
- 研究担当者:池田達哉、長嶺 敬、福岡浩之、矢野 博
- 発表論文等:1)池田達哉ら (1999) 育種学研究1(別2):213.
2)池田達哉ら (2000) 育種学研究2(別2):403.
3)T. M. Ikeda et al. (2002) Theoret. Appl. Genet.104:680-687.
