背景とねらい
果樹類では、品種の開発期間を大幅に短縮する技術としてだけではなく、育成権者の権利を保護しなければならない育成品種を判別する目的でも、DNAマーカーが開発されています。柑橘類やモモ、ナシ、リンゴといった主要な果樹では、すでに50を超えるDNAマーカーが開発され、200品種以上に適用されている例もあります。DNAマーカーをそれぞれの品種に適用して得られたマーカー型は一覧表に整理され、どのDNAマーカーが品種判別に有効であるか、統計的な方法で事前に評価します。こうした評価は、新しくDNAマーカーが追加されたり、適用された品種が追加されたりするごとに改めて行う必要があります。ところが、DNAマーカー数や適用した品種数が増えると、それに伴って計算量も増えてくるために、手軽なソフトで統計計算を行うことが困難になってきました。
そこで、簡単な操作で計算ができるソフトウエアMarkerToolKitを開発しました。
成果の内容・特徴
今回開発したソフトウエアMarkerToolKitは、品種判別用のDNAマーカーの有効性を評価するために、以下の計算機能を持っています。
- (1)SSRマーカーの数値表記について、アリル(対立遺伝子)の表記順を昇順に並べ替えて出力する。
- (2)全ての2品種の組合せで判別可能なマーカー数を出力する。
- (3)数値表記のマーカー型を文字表記に変換する。
- (4)アリル頻度、ヘテロ接合度、多型情報含有値など、各マーカーの品種判別力を評価する指標を出力する。
- (5)各マーカーのマーカー型の種類と頻度を出力する。
- (6)SSRマーカーのアリル間の数値の差の絶対値を出力する。
- (7)SSRマーカーの数値表記を相対表記に変換して出力する。
- (8)鵜飼の品種判別理論値(鵜飼, 2004)を出力する。
- (9)樹形図解析のためにアリルの存在を0/1形式で表記して出力する。
上記の9種類の計算のうち、既存のソフトウエアにはない(2)、(7)、(8)の3つの計算について紹介します。
1.既存のDNAマーカーでは判別できない品種の組み合わせが、簡単にわかります
例えば表1に示したような8種類の品種群について5種類のDNAマーカーを使って調べたデータをMarkerToolKitに入力すると、図1のような結果が得られます。図1で、白抜きで0と示されている品種の組み合わせでは、その2品種を判別することができるDNAマーカーが0個である、すなわち、そのようなDNAマーカーがひとつも存在しないことを示しています。そして、0となった品種の組み合わせを判別できるような新たなDNAマーカーを開発しなければならないことがわかります。
表1において、品種V1と品種V8および品種V4と品種V7のマーカー型を比べると、すべてのマーカー型が一致しているため、図1では、それぞれの品種の交点の数字が0と出力されます。
表1 マーカー型一覧表の例(SSRマーカーの場合)
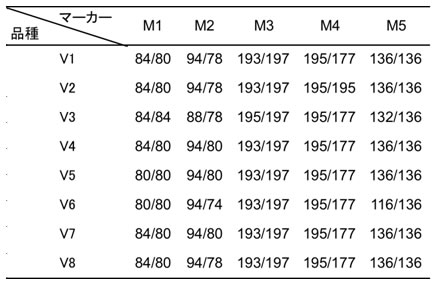
注)数値はSSRマーカーの塩基配列の長さを表す。
異なる数値は多型を示す。2倍体生物の場合、数値は2つになる。
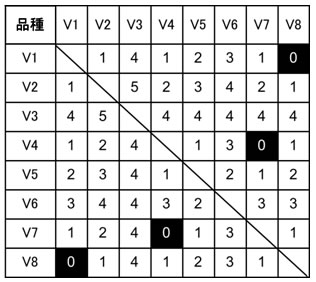
図1 表1についてすべての2品種の組合せにおける判別可能なマーカー数を総当りの表の形式で出力した結果
2.SSRマーカーの数値表記を相対表記に変換して出力します(表2)

SSRマーカーは、これまで、表1のような2つの数字で表記がなされてきました。これは、SSRマーカーが塩基配列の断片の長さによって、品種を判別する
という仕組みを利用しているため、塩基配列の断片の長さをそのまま表記するのが、これまでの表記法だったからです。しかし、最近になって、塩基配列の断片
の長さそのものよりも、ある断片の長さを基準にして、他の断片の長さを基準の断片の長さとの差を示す方法に国際的に統一されることになりました。ここで
は、前者を絶対表記、後者を相対表記と呼んでいます。MarkerToolKitは表1のような絶対表記のマーカー型一覧表を入力すると表2のような相対
表記に変換して出力します。MarkerToolKitでは、表1の最上行の品種V1のマーカー型の小さい方の数字を0として、それ以下の行の品種のマー
カー型をその差で表すことにしています。
例えば、表1のDNAマーカーM1では、品種V1のマーカー型の絶対表記は84/80ですから、表2の相対表記では、小さい方の数値の80を0とし、
84は+4と表すことになり、0/+4という表記になります。数値の順に意味はないので、MarkerToolKitではすべて昇順にならべて出力するこ
とにしています。
3.品種判別が可能かどうかの指標となる「鵜飼の品種判別理論値」を出力します(表3)
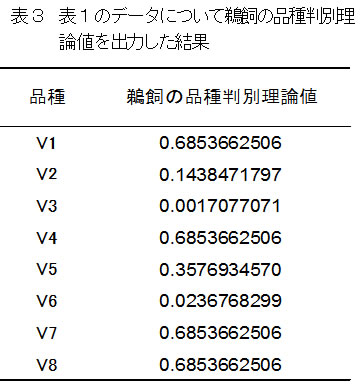
品種の権利保護を主張する場合、確実に該当品種であることを証明する必要があります。品種判別理論値は、DNAマーカーによって、他の品種と区別ができるかどうかの指標です。
品種判別理論値の値が0に近いV3のような品種ほど、表1のDNAマーカーによって、他の品種と確実に区別できることを示しています。反対に、品種判別理論値が1に近いV1、V4、V7、V8のような品種では、区別できないことになります。
品種判別理論値を0に近づけるためには、さらにDNAマーカーを増やして評価する必要があります。
今後の展望
Windowsや、Excelの対応バージョンは、順次拡大していく予定です。
用語解説
- DNAマーカー:DNA塩基配列の品種間での違いをもとに、ゲノム上の標識(目印)としたもの。染色体上の遺伝子内あるいはその近傍の塩基配列に違いがあり、その違いを識別できれば、DNAマーカーとして利用できる。DNAマーカーによって、特定の遺伝子を含む染色体のある領域が親から子へ受け継がれたかどうか検定できる。
- SSRマーカー:DNAマーカーの種類のうちのひとつ。ゲノム上の塩基配列の短い繰り返し部分の長さの違いを利用して品種を判別するDNAマーカーである。このためSSRマーカーは数字で示される。2倍体の生物の場合、80/84のように2つの数字で示される。多型性が高いので品種判別に有効である。
- アリル(対立遺伝子):生物を集団で見るとき、染色体上の相同な遺伝子座を占める遺伝子に複数の種類がある場合の、それぞれの遺伝子のこと。
- アリル頻度:生物を集団で見るとき、アリルの関係にある複数の種類の遺伝子のそれぞれが、集団内に存在する頻度。
- ヘテロ接合度:生物を集団で見るとき、遺伝子座におけるヘテロ接合体の割合。2倍体の生物の場合、父方と母方の2本の染色体があり、相同な1組の遺伝子座が存在する。その二つの遺伝子座を同じ種類の遺伝子(アリル)が占めている場合をホモ接合体、異なる種類の遺伝子(アリル)が占めている場合をヘテロ接合体という。ヘテロ接合度が高いほど、DNAマーカーとしての判別力が高い。
- 多型情報含有値:ヘテロ接合度と同様に、ヘテロ接合性を評価する指標のひとつ。この値が高いほどDNAマーカーとしての判別力が高い。
- 鵜飼の品種判別理論値:ある作物で、品種未知のサンプルが現れたとき、表1に示された品種の中に品種未知のサンプルと全く同一のマーカー型を示す品種が1品種以上存在する確率(鵜飼, 2004)。




