プレスリリース
DNAマーカーによる果樹育種の加速化
- おいしく、食べやすく、作りやすい果物の育成に向けて -
要約
果樹では、大果で食味に優れたおいしい品種、種なしで食べやすい品種、病気に強く栽培しやすい品種など、 消費者と生産者の双方にメリットのある品種が求められています。しかし、交配して得た種から開花・結実までに 数年間かかるため、新しい品種の育成には十数年以上の長い年月が必要です。 また、それらの栽培にも広大な面積を必要とするため、多くの個体を扱うことができません。
今回、果樹研究所では、ナシ、カンキツ、カキについて、以下のような重要形質を持つ個体を簡単に検出できる 有用なDNAマーカー*を開発しました。
- ナシの黒星病抵抗性及び黒斑病抵抗性。
- カンキツの種なし性及びカンキツトリステザウイルス抵抗性。
- カキの甘ガキ性。
これらの成果を利用すれば、幼苗の段階でそれぞれの形質について検定と選抜を行えることから、 果樹の新品種育成を大幅に効率的に進めることを可能にしました。
*DNAマーカー:
DNA塩基配列の品種間での違いを識別することで、 ゲノム上の標識(目印)としたもの。染色体上の遺伝子内あるいはその近傍の塩基配列に違いがあり、その違いを識別できれば、 DNAマーカーとして利用できる。DNAマーカーによって、特定の遺伝子を含む染色体のある領域が親から子へ受け継がれたかどうか検定できる。
詳細情報
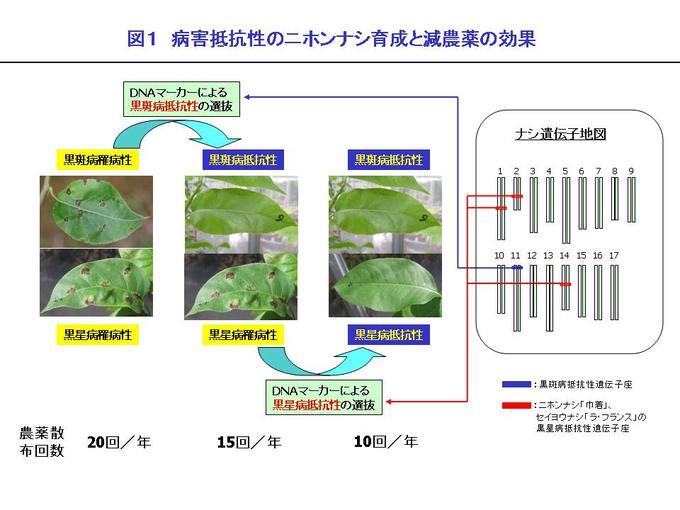
1.ナシの病害抵抗性
ニホンナシの栽培では、黒星病と黒斑病が最も重要な病気(病害)で、特に黒星病に抵抗性を持つ栽培品種はありません。 両方の病気に弱い品種では年間約20回の農薬散布を必要とし、一つの病害抵抗性品種では約5回の農薬散布を削減でき、 両方の抵抗性を持つことで農薬散布を半数に削減することが可能です。 私たちは、在来ニホンナシ品種「巾着」やセイヨウナシ品種「ラ・フランス」の黒星病抵抗性の遺伝子地図上の位置を 特定するとともに、抵抗性を持つ個体を簡単に検出できるDNAマーカーを多数取得しました。また、黒斑病抵抗性に連鎖する マーカーも取得することができました。これらのマーカーを使って効率的に有望な個体の選抜を行い、複数の病害抵抗性遺伝子を 持った新しい品種の育成を進めています(図1)。
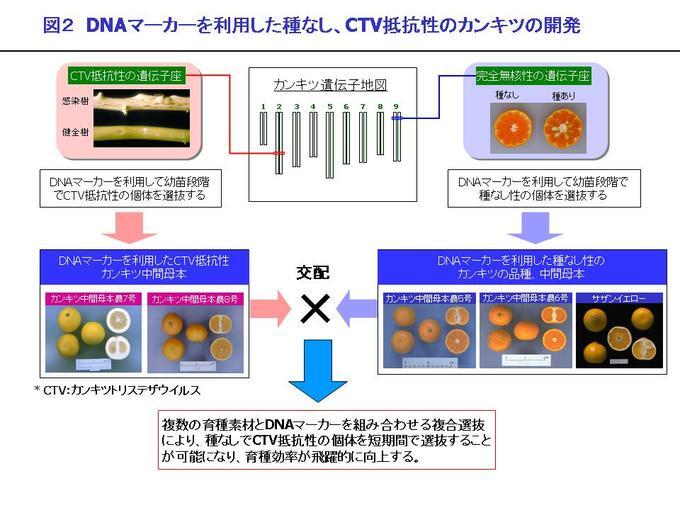
2.カンキツの種なし性及びカンキツトリステザウイルス抵抗性
カンキツでは、種なし化による食べやすさと病気に強く栽培しやすい特性を持つ品種が求められています。 在来のカンキツ品種「無核紀州」は、種なし性(無核性)遺伝子を持っており、交配するとその子供の約半数が種なしになります。 また、カンキツと近縁のカラタチは、世界的な重要病害であるカンキツトリステザウイルス(CTV)に対する抵抗性遺伝子を 持っています。私たちは静岡大学との共同研究により、カンキツの遺伝子地図を作成して「無核紀州」が持つ種なし性を 第9連鎖群に、カラタチのCTV抵抗性を第2連鎖群に位置づけ、それぞれのDNAマーカーを開発しました。 種なし性の特性を持つ育種素材とCTV抵抗性を持つ親を交配して得られた種子から幼苗段階に両方の特性を検出できるDNAマーカー による複合選抜を行うことにより、種なし性とCTV抵抗性を兼ね備えた新品種の効率的育成を進めています(図2)。
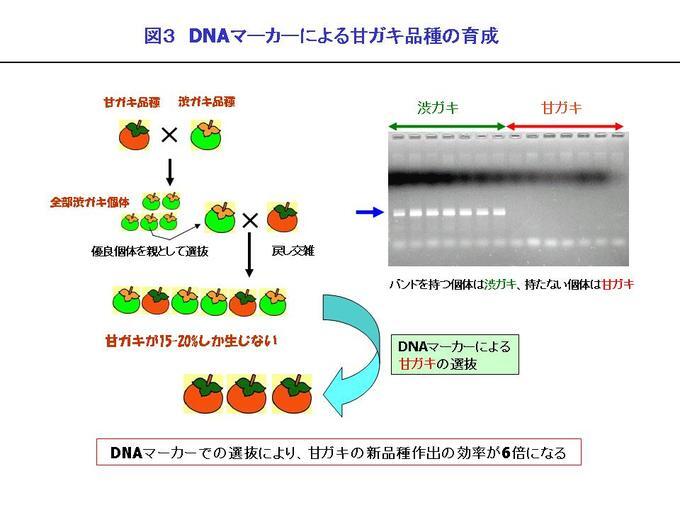
3.カキの甘ガキ性
カキには甘ガキと渋ガキがあり、甘ガキは果実に渋みが無い種類です。1,000を越える日本のカキ品種の中で、安定して甘ガキになるものは、 ごく少数しかありません。この甘ガキ性は遺伝的に劣性であるため、甘ガキ品種と渋ガキ品種を交配した世代では、 甘ガキの子供は生まれませんが、子供に繰り返して甘ガキ品種を交配すると、その次の世代では15~20%程度が甘ガキになります。 私たちは京都大学との共同研究により、甘ガキ性を識別するDNAマーカーを開発しました。種子から発芽した若い葉のDNAを調べることによって、 甘ガキと判定された個体だけを育てれば良いので、品種改良は大幅な効率化が可能です。現在、交配で作出した約3,000個体について、 DNAマーカーによる甘ガキの選抜を進めています(図3)。
なお本研究成果は、農林水産省委託プロジェクト研究「有用遺伝子活用のための植物(イネ)・動物ゲノム研究―DNAマーカーによる 効率的な新品種育成システムの開発」で得られたものです。
用語説明
遺伝子地図
染色体上の遺伝子やDNAマーカーの位置を示した地図。