短期間の流行における口蹄疫ウイルスのゲノム変異動態を解明
要約
口蹄疫ウイルス2010年宮崎分離株104株のゲノム全長解析により、短期間流行時のウイルス遺伝子置換率を算出し、変異の多いアミノ酸領域を特定した。
- キーワード:口蹄疫ウイルス、ゲノム解析、遺伝子変異、次世代シークエンサー
- 担当:動物衛生研究部門・越境性感染症研究領域・海外病ユニット
- 代表連絡先:電話029-838-7937
- 分類:研究成果情報
背景・ねらい
口蹄疫は偶蹄類動物において最も伝染力の強い病気である。口蹄疫ウイルスはゲノム複製時における変異率が高く、その結果、幅広い遺伝子多様性を示す。蔓延地域においては抗原変異株や宿主特異性を持った変異株も確認されている。本研究では、口蹄疫ウイルスの遺伝子が短期間の流行でどのように変異するかを明らかにするため、流行が3ヶ月以内に抑えられた2010年宮崎県での発生症例のうち異なる時期、場所、動物種から分離されたウイルスのゲノム全長を比較解析した。
成果の内容・特徴
- 次世代シークエンサーを用いて、2010年の口蹄疫発生292症例のうち、異なる日にち、場所、動物種の材料から分離されたウイルス104株のゲノム全長配列を決定した。
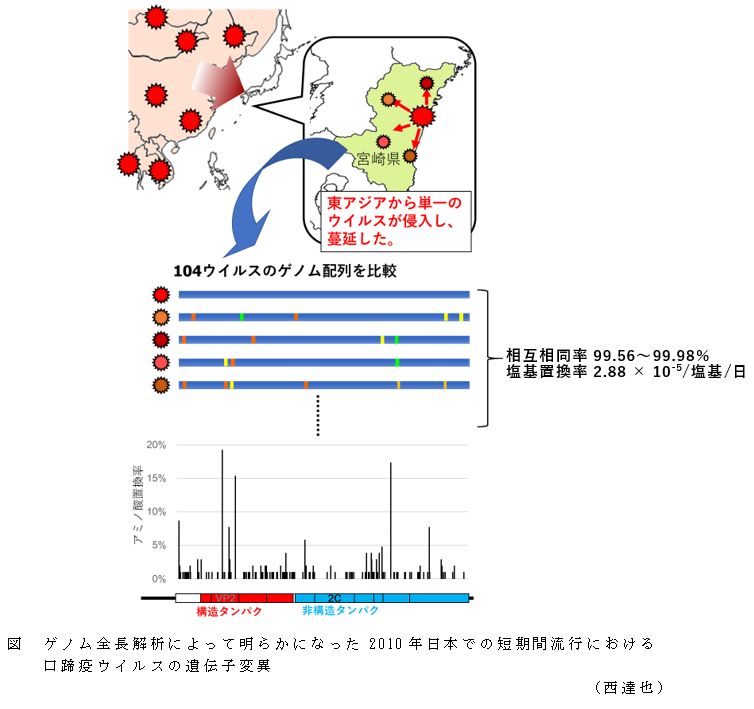
- 海外のウイルスのゲノム全長配列と共に作成した分子系統樹により、104株は一つのグループに分類されたことから、2010年の宮崎での口蹄疫発生時に単一のウイルスが侵入・蔓延したことがわかる。
- 104株のゲノム全体の相同性は99.56%~99.98%であり、完全に一致する配列はない。各ウイルス株の塩基配列と採材日から、塩基置換率は2.88 × 10-5/塩基/日であり、自然変異によるものと考えられる。迅速な防疫対応により発生規模を限局できたことで、性状の大きく異なるウイルスによる発生を防ぐことが出来たものと考えられる。
- 各アミノ酸の変異割合の解析結果、ウイルスの外殻タンパク質を構成するVP2は変異が集中して遺伝的に多様であり、非構造タンパク質である2Cには変異が少なく遺伝的に安定していることがわかる。
成果の活用面・留意点
- 次世代シークエンサーを用いて国内発生口蹄疫ウイルスのゲノム全長を迅速に決定し、海外のウイルスと系統解析することでそれらの起源を明らかにすることは、適切な防疫対策を講じる一助となる。
- 流行の時間枠中におけるウイルスの変異の動態を解明することで、より保存性の高いウイルス遺伝子領域をターゲットにした診断法の開発が期待される。
具体的データ
その他
- 予算区分:委託プロ(食の安全・動物衛生プロ)
- 研究期間:2013~2017年度
- 研究担当者:西達也、山田学、深井克彦、嶋田伸明、森岡一樹、菅野徹、山川睦
- 発表論文等:Nishi T. et al. (2017) Vet. Microbiol. 199:62-67
