コムギ縞萎縮病抵抗性遺伝子領域の高効率選抜マーカー
要約
北海道の秋播きコムギ品種の「ゆめちから」由来のコムギ縞萎縮病(WYMV)抵抗性遺伝子領域(Q.Ymym)に強連鎖する共優性マーカーであり、効率的な抵抗性系統の選抜が可能である。
- キーワード:コムギ縞萎縮病、ゆめちから、Q.Ymym、ゲノム情報、選抜マーカー
- 担当:次世代作物開発研究センター・基盤研究領域・育種法開発ユニット
- 代表連絡先:電話 029-838-7930
- 分類:研究成果情報
背景・ねらい
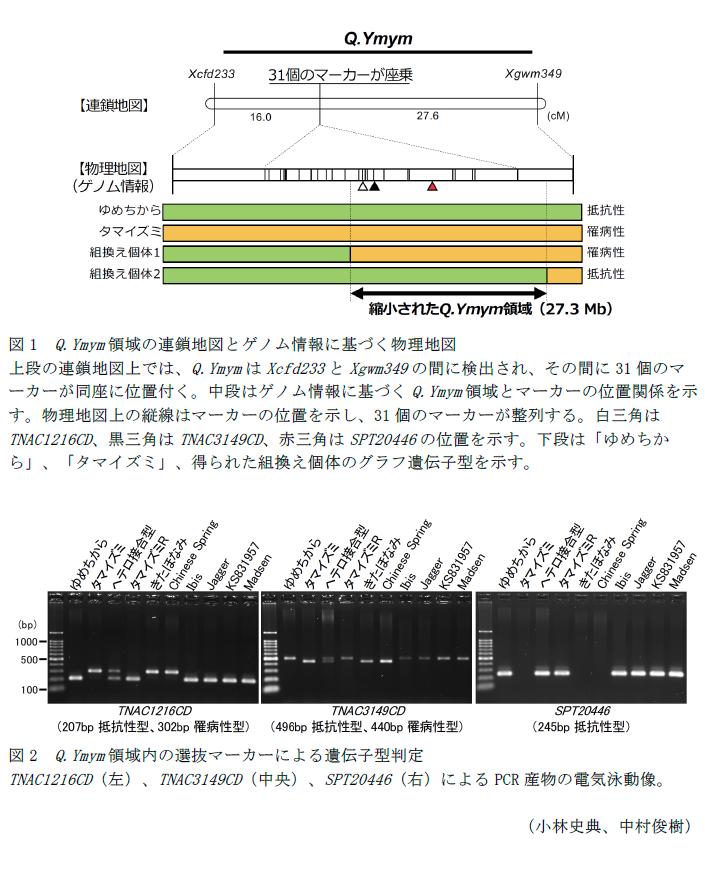
コムギ縞萎縮病は、国内の小麦生産にとって深刻な被害をもたらす。本病の防除対策として、抵抗性品種の作付けが最も効果的であるため、各地に適応した抵抗性品種の育成が求められている。北海道の秋播きコムギ品種の「ゆめちから」はコムギ縞萎縮病に対して高度な抵抗性を示し、2D染色体上に抵抗性遺伝子領域Q.Ymymを持つ。Q.Ymym領域は2D染色体長腕の末端部に位置し、その推定サイズは68.5Mb(6850万塩基対、2D染色体全体の10.5%に相当)と推定されている。複数の研究結果は、このQ.Ymym領域は遺伝的組換え頻度が非常に低いと報告している。既に、同領域選抜のための合計31個の連鎖マーカーが開発されているが(図1)、そのほとんどが優性マーカーであり、それらの位置関係も判明していない。そこで本研究では、最新のコムギ全ゲノム情報を利用してQ.Ymym領域のゲノム配列構造を調査し、31個の連鎖マーカーの位置関係を調べ、抵抗性遺伝子をより効率的に導入選抜するためのマーカーの選択、あるいは開発を行う。
成果の内容・特徴
- 「ゆめちから」を母本とした派生個体集団の中から既知のQ.Ymym領域内で組換えを起こした個体を取得し、それらの抵抗性の有無から、抵抗性遺伝子を含むQ.Ymym領域は、半分以下の27.3Mb(2730万塩基対)に絞り込まれる(図1)。
- 縮小したQ.Ymym領域内のゲノム配列は、参照ゲノム配列や罹病性品種の2D染色体上の同位置の配列と比べて相同性が低く、特殊なゲノム配列構造となっている。この配列変異は、スイスのコムギ系統「CH Campala Lr22a」の2D染色体上のハプロタイプブロックCで見られる配列構造と一致する。ハプロタイプブロックCの座乗位置はQ.Ymym領域と同一であり、「ゆめちから」と同じく遺伝的組換えが起きにくい。両者で見られるこのような配列変異が、遺伝的組換えを抑制する要因の一つと推察される。
- 参照ゲノム配列より既存の31連鎖マーカーの物理的座乗位置が判明し(図1)、その内の16個は縮小されたQ.Ymym領域内にある。その中でTNAC1216CDマーカーとTNAC3149CDマーカーを共優性化し、これらを用いることで遺伝子型の判別が容易になる(図2)。
- 「CH Campala Lr22a」のハプロタイプブロックCに存在する特異的遺伝子から開発されたPCRマーカー(SPT20446)は優性マーカーであるが、Q.Ymym領域の中央部に位置し(図1)、同抵抗性遺伝子型を識別できる(図2)。そのため、上記の2共優性マーカーによるQ.Ymym領域の導入選抜を補完する。
成果の活用面・留意点
- マーカーのプライマー配列とPCR条件は、Kobayashi et al.(2020)に記載されている。
- 成果情報「生麺の色の経時劣化との強連鎖を解消した新規コムギ縞萎縮病抵抗性母本」にて開発された新規抵抗性系統を交配母本に使用する際に、本研究で開発された選抜マーカーを利用することで、不良形質(高活性型ポリフェノール酸化酵素遺伝子)を持たない抵抗性個体の選抜が容易になり、スマート育種技術として展開できる。
具体的データ
その他
- 予算区分:交付金、競争的資金(科研費)
- 研究期間:2010~2019年度
- 研究担当者:小林史典、中村俊樹、小島久代、田中剛、齋藤美香、乙部千雅子
- 発表論文等:Kobayashi F. et al. (2020) Plant Breed. 139:93-106
