高精度DNAマーカーの開発と解析をMS Excel上でサポートするソフトウェアGUGSの開発
要約
DNAマーカーの開発と解析のためのツールGeneral Utilities for Genotyping Study (GUGS)の利用により、遺伝情報の変換と評価、親子関係の推定や連鎖解析、DNAマーカー開発のための塩基配列の変換や評価などの一連の解析をMS Excel単体で実行することで作業性が向上する。
- キーワード:DNAマーカー、遺伝子型情報、親子推定、遺伝統計解析、集団遺伝学
- 担当:果樹茶業研究部門・カンキツ研究領域・カンキツゲノムユニット
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
高精度なDNAマーカーによる遺伝子型解析は幅広い分野で利用されており、DNAマーカーと品種の2次元データを整理し、譲渡する目的で表計算ソフトウェアMS Excelがよく利用されているが、それ自身には遺伝子型データを扱う機能はない。DNAマーカーの設計や遺伝子型データの解析では目的ごとに異なるソフトウェアツールを使用するが、用いるツールのインストールやマニュアルの参照に加え、ツールごとに異なるデータ形式の変換やデータの取り出し、結果の取り込みなどの煩雑な作業に習熟することが必要で、意図しないミスが起きたり解析全体の律速となっている。そこで、DNAマーカーの設計から、得られる遺伝子型データの解析で利用される主要な機能をMS Excel上で実行するソフトウェアを開発し、DNAマーカーの設計から解析への工程のスループット向上を図る。
成果の内容・特徴
- GUGSは遺伝子型変換、基本解析、連鎖解析、データセット解析、遺伝子型頻度解析、親子推定などの遺伝解析と、DNAマーカー開発を支援する塩基配列操作においてよく利用される機能をMS Excel上で実行する110以上の関数で構成されており(表1)、2倍体ゲノムを有する植物、動物のDNAマーカー分析に幅広く対応することができる。
- 共優生のSSRやSNPマーカー遺伝子型に加え、連鎖解析で利用される一文字の遺伝子型記号や、DNA、RNAの塩基配列をMS Excel上で直接取り扱うことができる。GUGSの機能は関数として実装されており、MS Excelにビルトインされている関数とも任意に組み合わせて解析することができるため、解析の自由度が高い。
- DNAマーカーの設計からデータ解析までの主要な操作を単一のプラットフォーム上で実行することが可能となり、作業をMS Excel上で一貫して行うことでデータの変換や他のソフトウェアとのやり取りの過程で発生するミスを防止し、短時間で作業を完了することができる。
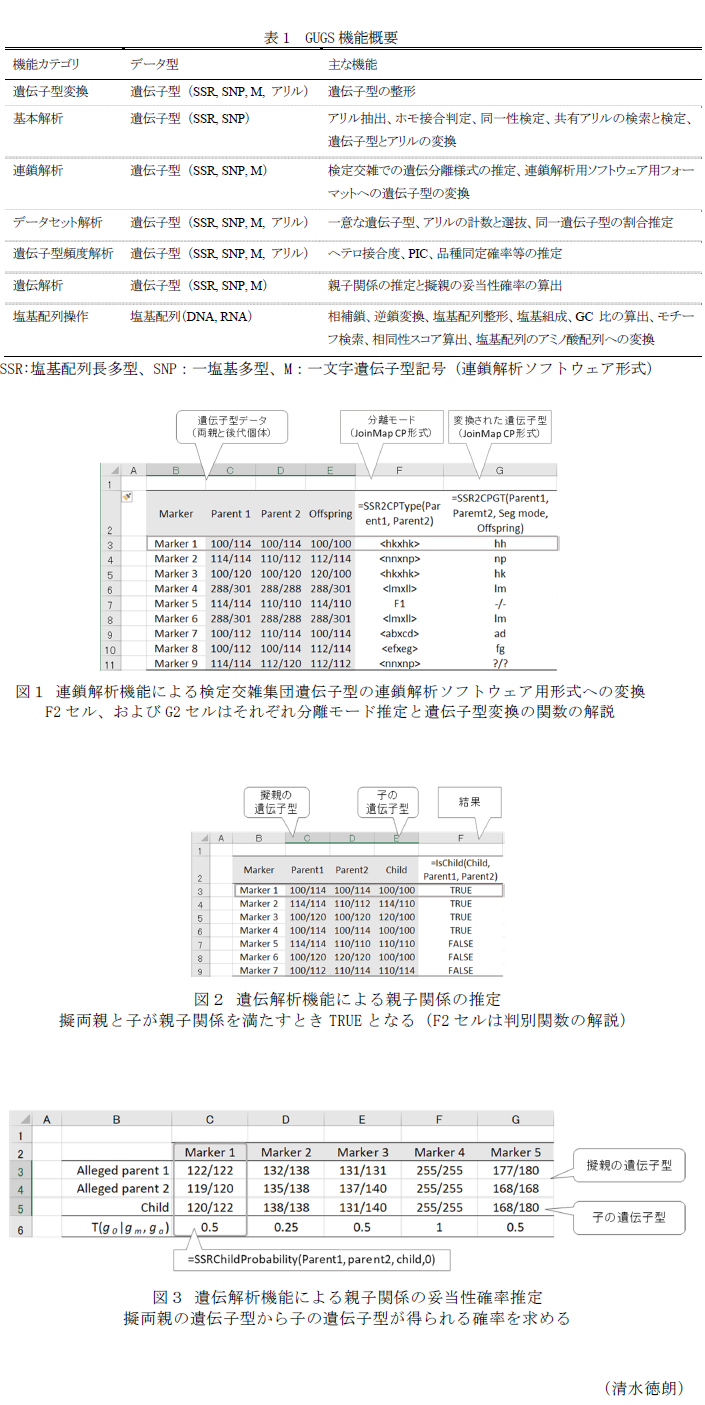
- 連鎖解析機能では、与えられた遺伝子型の分離モード推定や連鎖解析ソフトウェアJoinMap™やMapMakerの入力フォーマットへの遺伝子型変換をサポートしている(図1)。遺伝子型頻度解析では与えられたデータセットからヘテロ接合度やDNAマーカーの分析能力指標などを推定することができる。遺伝解析では与えられた遺伝子型値をもとに親子関係の推定(図2)、ならびに推定された親子関係の妥当性確率を求めることができる(図3)。
成果の活用面・留意点
- DNAマーカーの設計からデータ解析までの主要な操作を単一のプラットフォーム上で実行することが可能となり、DNAマーカーを取り扱う多様な解析の作業性が劇的に改善される。
- GUGSはMS Excel VBAで動作し、MS Excel 2010、2013、2016およびMS Office 365での動作を確認している(MS WindowsおよびMac OS)。使用にはマクロ実行を許可する必要がある。
- マニュアルとともにGPLv3で https://github.com/tokurou/GUGS にて公開しており、拡張性が高い。
具体的データ
その他
- 予算区分:交付金、競争的資金(科研費)、その他外部資金(SIP「スマートバイオ産業・農業基盤技術」)
- 研究期間:2015~2020年度
- 研究担当者:清水徳朗
- 発表論文等:
- Shimizu T. (2021) General Utilities for Genotyping Study (GUGS): A Comprehensive Application in Genotype and Sequence Data Manipulation. Japan Agricultural Research Quarterly (JARQ) 55(4):333-339.
- Shimizu T. et al. (2016) Hybrid Origins of Citrus Varieties Inferred from DNA Marker Analysis of Nuclear and Organelle Genomes. PLOS One 11(11): e0166969.
- 清水徳朗、他(2020)静岡県沼津市戸田の自生タチバナ群落の多様性解析とその維持機構の推定.園学研.(Hort. Res. (Japan)) 19 (2):141-149.
