全ゲノム解析により確認された新規オルソブニャウイルス
要約
次世代シーケンス解析とウイルスゲノムの末端配列を解析する手法の改良により、1980年代に分離された未同定ウイルスのゲノムの全塩基配列を決定する。この未同定ウイルスは、オルソブニャウイルス属の新たなウイルス種であることが明らかになり、Taniyama virusと命名する。
- キーワード : アルボウイルス、媒介節足動物、ヌカカ、新規ウイルス、次世代シーケンス解析
- 担当 : 動物衛生研究部門・越境性家畜感染症研究領域・疫学・昆虫媒介感染症グループ
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
九州・沖縄地域では、牛や媒介節足動物(ヌカカ)から数多くの節足動物媒介ウイルス(アルボウイルス)が得られているが、既知配列に基づくPCR法などでは同定できないものが見出される。近年、新規ウイルスの登録にあたっては、ゲノム全長の塩基配列情報が求められているが、オルソブニャウイルス属のように一本鎖RNAをゲノムに持つウイルスでは、ゲノム末端配列が高次構造を採ることから、塩基配列の決定が困難であった。そこで、本研究では次世代シーケンス解析と改良した末端配列解析法を組み合わせることにより、未解析のウイルスのゲノム全長の塩基配列を決定し、新規のアルボウイルスの同定を効率的に実施する。
成果の内容・特徴
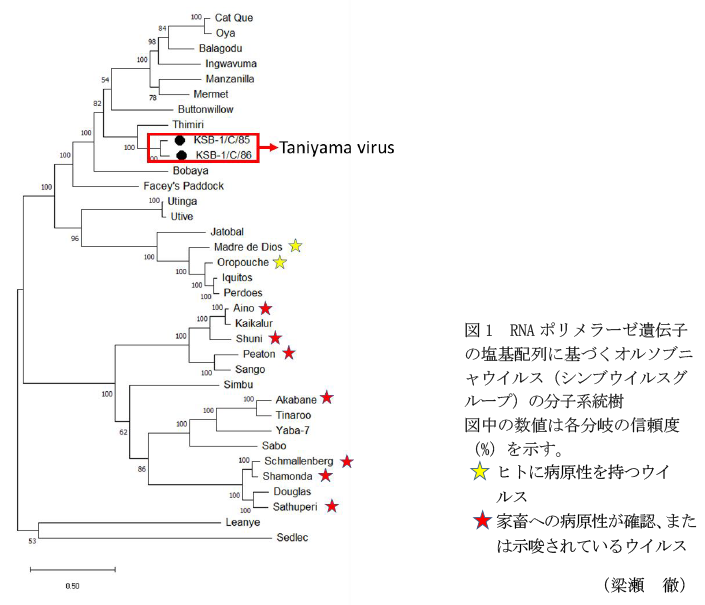
- 次世代シーケンス解析では、1985年と1986年にヌカカから分離された未解析のウイルス(KSB-1/C/85、KSB-1/C/86)の末端領域を除くゲノムの塩基配列は、これらのウイルスがオルソブニャウイルス属のシンブウイルスグループに属する新規ウイルスであることを示す(データ省略)。
- ウイルスゲノム複製の過程で、感染細胞内に出現する長鎖の二本鎖RNA(ウイルスのゲノムRNAおよびその相補的RNA)を使用することにより、ウイルスゲノムRNAと相補的RNAの3'末端領域の完全長cDNAを効率よく同時に増幅し、塩基配列を決定することができる。相補的RNAの3'末端領域の逆相補配列は、ウイルスゲノムRNAの5'末端領域の配列と一致するため、高次構造の影響によりcDNAの増幅が難しいウイルスゲノムRNAの5'末端領域の解析を実施する必要がない(データ省略)。
- RNAウイルスの分類に用いられるRNAポリメラーゼ遺伝子の分子系統解析の結果は、新規ウイルスはオルソブニャウイルス属シンブウイルスグループのうち、人に病気を起こすウイルスと比較的近縁であることを示す(図1)。国際ウイルス分類委員会の基準に照らし合わせると、既知ウイルスのRNAポリメラーゼ遺伝子の配列との違いから、新規ウイルスは新種と判断され、Tanaiyama orthobunyavirus 種(新種)のTaniyama virusとして、全ゲノム塩基配列情報とともに登録する。
成果の活用面・留意点
- Taniyama virusに対する抗体の検出系や遺伝子検査系を作出し、人や家畜の疾病との関連の有無を継続的に監視する必要がある。
- 一本鎖RNAウイルスが感染した細胞内で形成される、長鎖二本鎖RNAを用いた効率的なゲノム末端配列の決定は、末端配列が関与するウイルスゲノムの複製やゲノムRNAの粒子内への取り込み、あるいは転写調節などの分子メカニズムの解明を容易にする。
具体的データ
その他
- 予算区分 : 農林水産省(包括的レギュラトリーサイエンス研究推進委託事業:家畜の伝染病の国内侵入と野生動物由来リスクの管理技術の開発)
- 研究期間 : 2021~2022年度
- 研究担当者 : 梁瀬 徹、室田勝功、須田遊人
- 発表論文等 : Yanase T. et al. (2023) Arch. Virol. 168:67
