ナノポアシーケンスによる豚熱ウイルス国内流行株と経口生ワクチン株の迅速識別法
要約
ナノポアシーケンサーを用いて豚熱ウイルス国内流行株と経口生ワクチン株の塩基配列を決定し、ウイルス株を識別する方法である。従来法と比較して、識別までの時間を2時間以上短縮でき、検査の迅速化に有効である。
- キーワード : 豚熱、遺伝子診断、経口生ワクチン、ナノポアシーケンス、野生イノシシ
- 担当 : 動衛研部門・人獣共通感染症研究領域・新興ウイルスグループ
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
我が国では、2018年9月に26年ぶりとなる豚熱の発生が確認された。また、野生イノシシにおける豚熱の発生地域は拡大し続けている。野生イノシシにおける豚熱検査は、「豚熱に関する特定家畜伝染病防疫指針」に沿って実施されており、血清、扁桃、脾臓または腎臓組織からコンベンショナル、または、リアルタイムRT-PCR法を用いて豚熱ウイルス(CSFV)核酸を検出している。コンベンショナルRT-PCR法の標的増幅領域は、CSFV株間で高度に保存されているため、高感度にCSFVを検出できるものの、①CSFV国内流行株、②野生イノシシへの免疫付与に使われている経口生ワクチン株(C株)、③飼養豚用の弱毒生ワクチン株(GPE-株)の3株の識別はできない。従って、ウイルス株を同定するためにはRT-PCR産物(アンプリコン)の塩基配列を決定する必要がある。
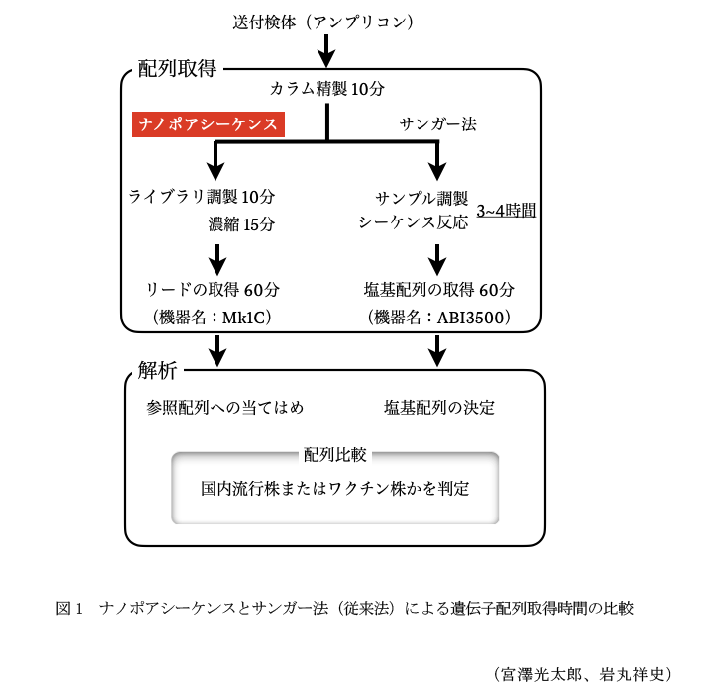
ナノポアシーケンスでは従来のシーケンス反応が不要であり、これまでよりも迅速・簡便に塩基配列を決定できることが示唆される。本研究では、ナノポアシーケンサーを用いてCSFVのアンプリコンの塩基配列を決定し、サンガー法に基づく現行法と同等の精度で野外株とワクチン株の識別が可能か検証する。
成果の内容・特徴
- 1検体当り10 ng以上のPCR産物を含むライブラリを調製後、12検体を同時に測定用カセットに添加し、ナノポアシーケンスを実施する(使用機器:MK1C)。得られたリードを基に各検体のコンセンサス配列を取得する。ナノポアシーケンスの結果から得たコンセンサス配列は、サンガー法で得られた塩基配列と完全に一致し、野外株かワクチン株かを判定可能である。
- 計29検体のPCR産物の塩基配列をサンガー法とナノポアシーケンスの両方で決定し、比較する。野外株とワクチン株の識別に必要な領域の塩基配列は1検体を除いて完全に一致するため、ナノポアシーケンスを用いても野外株とワクチン株を識別できる。
- 本法では、現行のサンガー法(使用機器:ABI3500)と同等の塩基精度を保ちながら、少なくとも検査時間を2時間短縮可能である(図1)。
成果の活用面・留意点
- ナノポアシーケンサーを用いてCSFV国内流行株と経口生ワクチン株を識別するには、参照塩基配列に複数のリードを照合する必要がある。そのため、1検体を1個の測定用カセットに注入するか、核酸量を揃えた複数検体を1個の測定用カセットに注入し、各検体から十分なリード数を得ることが重要である。
- 豚熱のみならず、将来的に各都道府県の家畜保健衛生所において病原体ゲノム解析の必要性が高まれば、費用対効果に優れた遺伝子解析ツールとしてナノポアシーケンスは期待できる。
具体的データ
その他
- 予算区分 : 交付金、農林水産省(包括的レギュラトリーサイエンス研究推進委託事業: CSFの新たな総合的防除技術の開発)
- 研究期間 : 2020~2021年度
- 研究担当者 : 宮澤光太郎、松浦裕一、池田圭吾、岩丸祥史
- 発表論文等 : 宮澤ら(2022)日本獣医師会雑誌、75(5):e83-e90
