アジア型カンキツグリーニング病原細菌は少なくとも5グループに分類される
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
バクテリオファージ型DNAポリメラーゼ遺伝子領域の有無および塩基配列の比較により、日本分離株を含むアジア型カンキツグリーニング病原細菌は少なくとも5つのグループに分類される。日本分離株の一部は独自のグループを形成する。
- キーワード:カンキツグリーニング病、アジア型カンキツグリーニング病原細菌、集団構成、バクテリオファージ型DNAポリメラーゼ遺伝子領域
- 担当:果樹研・カンキツグリーニング病研究チーム、九州沖縄農研・暖地施設野菜花き研究チーム
- 代表連絡先:電話029-838-6544
- 区分:果樹・病害虫
- 分類:研究・普及
背景・ねらい
世界各地のカンキツ生産地で発生しているカンキツグリーニング病原細菌は、アジア型、アフリカ型およびアメリカ型の3種が知られている。しかしながら、日本を含んだアジア地域に発生しているアジア型カンキツグリーニング病原細菌(Candidatus Liberibacter asiaticus; Las)の詳細な遺伝的多様性は知られていない。そこで、幾つかの遺伝子領域を用いて遺伝的多様性を評価することで、Lasの集団構成および地理的な分布を明らかにする。
成果の内容・特徴
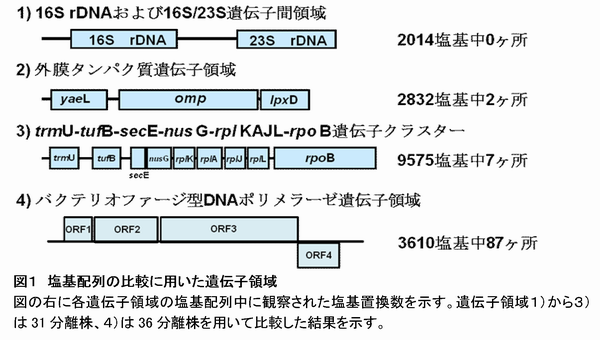
- 本研究で比較に用いた遺伝子領域は16S rDNAおよび16S/23S遺伝子間領域、trmU-tufB-secE-nusG-rplKAJL-rpoB遺伝子クラスター、外膜タンパク質遺伝子領域およびバクテリオファージ型DNAポリメラーゼ遺伝子領域(DNA poly)である。
- Lasの遺伝的多様性は、比較に用いる遺伝子領域によって大きく異なり、バクテリオファージ型DNAポリメラーゼ遺伝子領域(DNA poly)で最も高く、16S rDNAおよび16S/23S遺伝子間領域、trmU-tufB-secE-nusG-rplKAJL-rpoB遺伝子クラスターおよび外膜タンパク質遺伝子領域では殆どみられない(図1)。
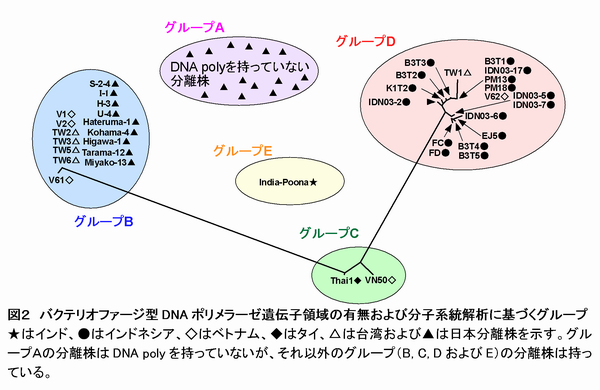
- Las分離株はDNA polyの有無により、DNA polyを持っていない集団(グループA)と持っている集団(グループB, C, DおよびE)に大別される。
- インドネシア、タイ、ベトナム、台湾および日本で採集した36分離株のDNA polyの塩基配列をもとに作成した分子系統樹により、これら諸外国産Las分離株は3集団(グループB, Cおよび D)を形成する(図2)。
- 既報のインド産India-Poona分離株のDNA polyの一部塩基配列の相同性は、本研究で塩基配列を決定した分離株と大きく異なることから、さらに1つの集団(グループE)を形成する。
- 日本国内で分離されたLas株は、DNA polyを持っていない集団(グループA)と持っている集団に大別され、DNA polyを持っている集団は全てグループBに含まれる(図2)。グループAは、日本分離株のみで構成される。
成果の活用面・留意点
- 新たにカンキツグリーニング病が発見された際に、Lasのバクテリオファージ型DNAポリメラーゼ遺伝子領域(DNA poly)の有無、塩基配列から、Lasの由来が推定でき、水際での防除に役立てることができる。
具体的データ
その他
- 研究課題名:気候温暖化等環境変動に対応した農業生産管理技術の開発
- 中課題整理番号:215a.5
- 予算区分:基盤、交付金プロ(温暖化適応)
- 研究期間:2007年~2009年度
- 研究担当者:冨村健太、宮田伸一、古屋典子、久保田健嗣、奥田充、岩波徹
- 発表論文等:Tomimura et al. (2009) Phytopathology 99(9):1062-1069
