ニホンナシから同定した新規レトロトランスポゾン
要約
ニホンナシから単離した8種類の新規レトロトランスポゾンは、約9kbpのコピア型の遺伝子配列を持ち、構造的に2種類に大別できる。蛍光 in situ ハイブリダイゼーション解析から、数十のコピーが染色体に散在する。
- キーワード:ニホンナシ、コピア型、レトロトランスポゾン
- 担当:果樹・茶・果樹ゲノム利用技術
- 代表連絡先:成果情報のお問い合わせ
- 研究所名:果樹研究所・品種育成・病害虫研究領域
- 分類:研究成果情報
背景・ねらい
レトロトランスポゾンは、可動遺伝因子の一種であり、多くの真核生物組織のゲノム内に普遍的に存在する。トウモロコシではゲノムの80%、コムギではゲノムの90%を占めており、植物では核DNAの主要成分となっている。自分自身をRNAに複写した後、逆転写酵素によってDNAに複写されてから新たな場所に挿入することで転移する。
レトロトランスポゾンはゲノム中に散在し、コピー数が多く、また転移してゲノムに挿入された後に安定に保持されるので、ゲノムワイドなDNAマーカーを作成するための候補配列として着目されている。バラ科果樹ではこれまでリンゴで1例報告があるだけでニホンナシではほとんど情報がなかったため、新規レトロトランスポゾンを同定し全長の塩基配列を決定する。
成果の内容・特徴
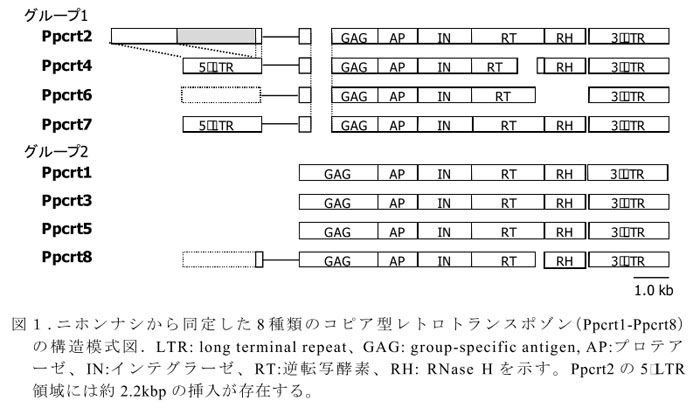
- ニホンナシ品種「巾着」のBACライブラリから、8種類の新規レトロトランスポゾンPpcrt1-Ppcrt8を同定し塩基配列を決定した。いずれも末端に長い反復配列を有するLTR型で、遺伝子の並び順からコピア型である(図1)。全長は約9kbで、その配列中には塩基の挿入や置換などの変異が観察され、GAG配列(group-specific antigen)中の142アミノ酸の欠失の有無から2つのグループに大別される。
- 得られたニホンナシのレトロトランスポゾンは、遺伝子配列の相同性から、イネのRIRE1やコムギのBARE-1と近縁である。
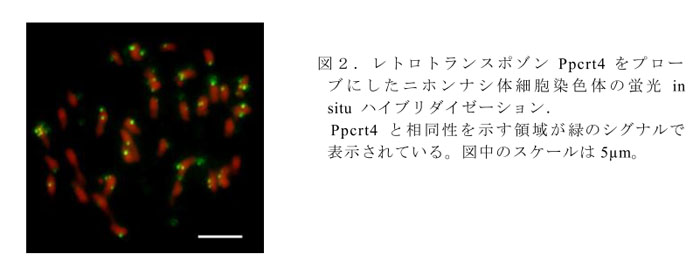
- 蛍光 in situ ハイブリダイゼーション(fluorescence in situ hybridization, FISH)(図2)とゲノミックサザン分析(データ省略)によるゲノム中のコピー数と存在位置の解析から、数十のレトロトランスポゾンが多くの染色体に散在する。
成果の活用面・留意点
- ニホンナシから同定した8種類のレトロトランスポゾンの塩基配列情報は、公的遺伝子データベースに登録済みである(アクセッション番号:AB550651-AB550658)。
- レトロトランスポゾンの塩基配列情報をもとに、ゲノムワイドなDNAマーカーを作成し、より高密度な連鎖地図作成に供することが可能である。
- レトロトランスポゾンの塩基配列情報をもとに、ナシ品種判別用のDNAマーカーを作成することが可能である。
具体的データ
(山本俊哉)
その他
- 中課題名:果樹におけるDNAマーカー育種のための高度基盤技術の開発
- 中課題番号:142g0
- 予算区分:交付金
- 研究期間:2008~2011年度
- 研究担当者:山本俊哉、金 會澤(契約職員等)、寺上伸吾、西谷千佳子、山本雅史(鹿児島大農)、松本 隆(生物研)、松山知樹(理化学研究所)
- 発表論文等:Kim H. et al. (2011) Tree Genetics & Genomes 7: 845–856.
