リンゴ全ゲノムをカバーする台木および野生種の連鎖地図
要約
リンゴ台木「JM7」とリンゴ属野生種のミツバカイドウ「サナシ63」の連鎖地図はほぼ全ゲノムをカバーし、多数のSSRマーカーを含む。このためリンゴ台木重要形質解析の基盤として用いることができる。
- キーワード:リンゴ台木、リンゴ属野生種、ゲノム、連鎖地図、SSRマーカー
- 担当:果樹・茶・リンゴ
- 代表連絡先:電話 029-838-6453
- 研究所名:果樹研究所・リンゴ研究領域
- 分類:研究成果情報
背景・ねらい
リンゴ台木と野生種を交雑してリンゴ台木の育種を行う際、リンゴ台木に由来するわい化性や挿し木発根性および野生種に由来する根頭がんしゅ病抵抗性等の重要形質の評価には圃場や温室において大規模な試験を長期間行う必要がある。これを効率化するためには、重要形質に連鎖するDNAマーカーを開発し、マーカー選抜によって試験対象個体を絞り込むことが有効である。しかしながら、重要形質の多くは量的形質であり、形質に連鎖するマーカーの獲得にはゲノムを広範囲にカバーする連鎖地図を構築しQTL解析を行うことが必須である。そこで、重要形質に連鎖するDNAマーカー開発の基盤とするため、全ゲノムをカバーする連鎖地図の構築を行う。また、ナシ亜科植物間で利用できるEST-SSRを新たに開発し、連鎖地図にマッピングする。
成果の内容・特徴
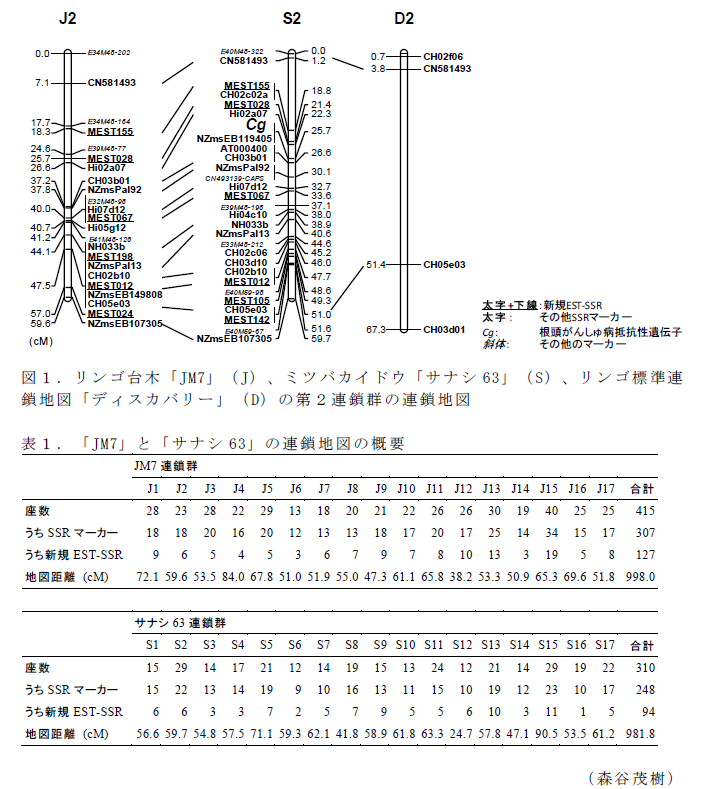
- リンゴ台木「JM7」とリンゴ属野生種ミツバカイドウ「サナシ63」の連鎖地図はいずれも17連鎖群で構成され、それぞれ415座、310座から成る。また、合計地図距離はそれぞれ998.0cM、981.8cMであり、平均マーカー間距離は2.4cM、3.2cMである(図1、表1)。また、「サナシ63」の第2連鎖群には根頭がんしゅ病抵抗性遺伝子Cgがマッピングされている(図1)。
- 連鎖地図のうち、SSRマーカーで得られる遺伝子座は「JM7」では307座、「サナシ63」では248座である(表1)。連鎖地図には、リンゴ発現遺伝子情報から新規開発されたEST-SSRマーカー137個を含んでおり、「JM7」と「サナシ63」の連鎖地図上にそれぞれ127座、94座存在する(表1)。
- 連鎖地図は共通のSSRマーカーをアンカーとしてリンゴ標準連鎖地図と対応付けることができる(図1)。各連鎖群の両端に位置するアンカーから、本連鎖地図がカバーするゲノム領域はリンゴ標準地図のカバー領域と同等であり、ほぼ全ゲノムをカバーしている。
成果の活用面・留意点
- 本連鎖地図はほぼ全ゲノムをカバーし、高密度にマーカーが座乗するため、量的形質であるリンゴ台木重要形質のQTL解析に用いることができる。
- 開発した137種類の新規EST-SSRマーカーの塩基配列情報はDDBJ、NCBI等の公的遺伝子データベースに登録済みである(アクセッション番号AB627185~AB627321)。
- 新規EST-SSRはナシやビワなど他のナシ亜科植物のゲノム解析にも利用可能であるが、分析にはDNAシーケンサーを用いる必要がある。
具体的データ
その他
- 中課題名:高商品性リンゴ等品種の育成と省力生産技術の開発
- 中課題番号:142e0
- 予算区分:交付金
- 研究期間:2008年~2012年度
- 研究担当者:森谷茂樹、岩波宏、古藤田信博、土師岳、岡田和馬、寺上伸吾、耳田直純(筑波大)、山本俊哉、阿部和幸
- 発表論文等:Moriya S. et al. (2012) Tree Genet. Genomes 8:709-723
